Prediksi Risiko Diabetes Tahap Awal#
Pendahuluan#
Diabetes mellitus merupakan salah satu penyakit kronis yang paling umum di seluruh dunia dan menjadi ancaman serius bagi kesehatan masyarakat. Penyakit ini terjadi ketika tubuh tidak mampu memproduksi atau menggunakan insulin secara efektif, sehingga menyebabkan kadar gula darah meningkat. Menurut data Organisasi Kesehatan Dunia (WHO), prevalensi diabetes terus meningkat setiap tahunnya, bahkan di usia yang semakin muda. Kondisi ini mendorong perlunya strategi pencegahan yang efektif, salah satunya dengan mendeteksi risiko diabetes sejak dini.
Prediksi risiko diabetes tahap awal memiliki peran penting dalam upaya preventif, karena memungkinkan individu dengan risiko tinggi untuk mengambil tindakan sebelum gejala berkembang lebih lanjut. Dengan kemajuan teknologi dan pemrosesan data, kini tersedia berbagai metode analitik dan algoritma kecerdasan buatan yang dapat digunakan untuk mengidentifikasi faktor risiko berdasarkan data klinis seperti usia, indeks massa tubuh (IMT), tekanan darah, kadar glukosa, dan riwayat keluarga.
Dalam proyek ini, dilakukan pengembangan model prediksi risiko diabetes tahap awal menggunakan pendekatan komputasi data. Tujuannya adalah untuk membantu meningkatkan kesadaran masyarakat akan pentingnya deteksi dini serta mendukung tenaga medis dalam pengambilan keputusan berbasis data. Dengan pendekatan ini, diharapkan angka kejadian diabetes dapat ditekan melalui tindakan preventif yang lebih tepat sasaran dan efisien.
Data Understanding#
Sumber Data#
Dataset diambil dari link dibawah ini:
https://archive.ics.uci.edu/dataset/529/early+stage+diabetes+risk+prediction+dataset
Salah satu dataset yang umum digunakan dalam pengembangan dan penelitian klasifikasi medis adalah Early Stage Diabetes Risk Prediction yang tersedia di UCI Machine Learning Repository. Dataset ini memuat parameter biomekanis yang diambil dari age, gender, polyuria, polydipsia, sudden_weight_loss, weakness, polyphagia, genital_thrush, visual_blurring, itching.
Tujuan#
Penelitian ini bertujuan untuk mengembangkan dan mengimplementasikan algoritma klasifikasi dalam rangka memprediksi risiko diabetes pada tahap awal berdasarkan gejala klinis yang umum dialami oleh pasien. Dengan memanfaatkan data seperti usia, jenis kelamin, dan berbagai gejala seperti poliuria, polidipsia, penurunan berat badan secara tiba-tiba, serta keluhan umum lainnya, sistem yang dikembangkan diharapkan mampu memberikan prediksi yang akurat terhadap kemungkinan seseorang mengalami diabetes.
Model klasifikasi ini diharapkan dapat menjadi alat bantu diagnosis awal yang cepat dan efisien dalam lingkungan medis, serta membantu tenaga kesehatan dalam melakukan deteksi dini dan pengambilan keputusan yang lebih tepat terkait risiko diabetes.
Integrasi Data#
untuk mengambil data agar dapat diolah, perlu untuk menginstall package yang telah disediakan oleh UCI Dataset. Instalasi dilakukan berguna untuk menarik data yang berasal dari UCI dataset agar dapat diolah. peritah untuk mengambil data dari UCI dataset dapat di lihat ketika menekan tombol import in python pada datase yang diinginkan dan ikuti perintah tersebut agar data dapat diambil dari UCI dataset. Contoh pengambilan data dari UCI dataset dapat dilihat pada gambar dan perintah berikut:
!pip install ucimlrepo
Requirement already satisfied: ucimlrepo in /usr/local/python/3.12.1/lib/python3.12/site-packages (0.0.7)
Requirement already satisfied: pandas>=1.0.0 in /home/codespace/.local/lib/python3.12/site-packages (from ucimlrepo) (2.2.3)
Requirement already satisfied: certifi>=2020.12.5 in /home/codespace/.local/lib/python3.12/site-packages (from ucimlrepo) (2024.8.30)
Requirement already satisfied: numpy>=1.26.0 in /home/codespace/.local/lib/python3.12/site-packages (from pandas>=1.0.0->ucimlrepo) (2.2.0)
Requirement already satisfied: python-dateutil>=2.8.2 in /home/codespace/.local/lib/python3.12/site-packages (from pandas>=1.0.0->ucimlrepo) (2.9.0.post0)
Requirement already satisfied: pytz>=2020.1 in /home/codespace/.local/lib/python3.12/site-packages (from pandas>=1.0.0->ucimlrepo) (2024.2)
Requirement already satisfied: tzdata>=2022.7 in /home/codespace/.local/lib/python3.12/site-packages (from pandas>=1.0.0->ucimlrepo) (2024.2)
Requirement already satisfied: six>=1.5 in /home/codespace/.local/lib/python3.12/site-packages (from python-dateutil>=2.8.2->pandas>=1.0.0->ucimlrepo) (1.17.0)
[notice] A new release of pip is available: 24.3.1 -> 25.1.1
[notice] To update, run: python3 -m pip install --upgrade pip
from ucimlrepo import fetch_ucirepo
# fetch dataset
early_stage_diabetes_risk_prediction = fetch_ucirepo(id=529)
# data (as pandas dataframes)
X = early_stage_diabetes_risk_prediction.data.features
y = early_stage_diabetes_risk_prediction.data.targets
data = early_stage_diabetes_risk_prediction.data.original
data.to_csv("early_stage_diabetes_risk_prediction.csv", index=True)
print(data.info()) #untuk menampilkan info fitur-fitur yang ada di tabel
print(data.head()) #untuk menampilkan 5 baris pertama
<class 'pandas.core.frame.DataFrame'>
RangeIndex: 520 entries, 0 to 519
Data columns (total 17 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 age 520 non-null int64
1 gender 520 non-null object
2 polyuria 520 non-null object
3 polydipsia 520 non-null object
4 sudden_weight_loss 520 non-null object
5 weakness 520 non-null object
6 polyphagia 520 non-null object
7 genital_thrush 520 non-null object
8 visual_blurring 520 non-null object
9 itching 520 non-null object
10 irritability 520 non-null object
11 delayed_healing 520 non-null object
12 partial_paresis 520 non-null object
13 muscle_stiffness 520 non-null object
14 alopecia 520 non-null object
15 obesity 520 non-null object
16 class 520 non-null object
dtypes: int64(1), object(16)
memory usage: 69.2+ KB
None
age gender polyuria polydipsia sudden_weight_loss weakness polyphagia \
0 40 Male No Yes No Yes No
1 58 Male No No No Yes No
2 41 Male Yes No No Yes Yes
3 45 Male No No Yes Yes Yes
4 60 Male Yes Yes Yes Yes Yes
genital_thrush visual_blurring itching irritability delayed_healing \
0 No No Yes No Yes
1 No Yes No No No
2 No No Yes No Yes
3 Yes No Yes No Yes
4 No Yes Yes Yes Yes
partial_paresis muscle_stiffness alopecia obesity class
0 No Yes Yes Yes Positive
1 Yes No Yes No Positive
2 No Yes Yes No Positive
3 No No No No Positive
4 Yes Yes Yes Yes Positive
Letakkan jika anda ingin data dalam bentuk csv
from google.colab import files
files.download('early_stage_diabetes_risk_prediction.csv')
---------------------------------------------------------------------------
ModuleNotFoundError Traceback (most recent call last)
Cell In[3], line 1
----> 1 from google.colab import files
2 files.download('early_stage_diabetes_risk_prediction.csv')
ModuleNotFoundError: No module named 'google'
Eksplorasi Data#
VIsualisasi Data#
display(data) #display dataset
| age | gender | polyuria | polydipsia | sudden_weight_loss | weakness | polyphagia | genital_thrush | visual_blurring | itching | irritability | delayed_healing | partial_paresis | muscle_stiffness | alopecia | obesity | class | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 40 | Male | No | Yes | No | Yes | No | No | No | Yes | No | Yes | No | Yes | Yes | Yes | Positive |
| 1 | 58 | Male | No | No | No | Yes | No | No | Yes | No | No | No | Yes | No | Yes | No | Positive |
| 2 | 41 | Male | Yes | No | No | Yes | Yes | No | No | Yes | No | Yes | No | Yes | Yes | No | Positive |
| 3 | 45 | Male | No | No | Yes | Yes | Yes | Yes | No | Yes | No | Yes | No | No | No | No | Positive |
| 4 | 60 | Male | Yes | Yes | Yes | Yes | Yes | No | Yes | Yes | Yes | Yes | Yes | Yes | Yes | Yes | Positive |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 515 | 39 | Female | Yes | Yes | Yes | No | Yes | No | No | Yes | No | Yes | Yes | No | No | No | Positive |
| 516 | 48 | Female | Yes | Yes | Yes | Yes | Yes | No | No | Yes | Yes | Yes | Yes | No | No | No | Positive |
| 517 | 58 | Female | Yes | Yes | Yes | Yes | Yes | No | Yes | No | No | No | Yes | Yes | No | Yes | Positive |
| 518 | 32 | Female | No | No | No | Yes | No | No | Yes | Yes | No | Yes | No | No | Yes | No | Negative |
| 519 | 42 | Male | No | No | No | No | No | No | No | No | No | No | No | No | No | No | Negative |
520 rows × 17 columns
Tampilkan banyaknya class#
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
from ucimlrepo import fetch_ucirepo
# Ambil dataset Early Stage Diabetes Risk Prediction
early_stage_diabetes_risk_prediction = fetch_ucirepo(id=529)
# Ambil fitur dan target
X = early_stage_diabetes_risk_prediction.data.features
y = early_stage_diabetes_risk_prediction.data.targets
# Gabungkan fitur dan target
data = pd.concat([X, y], axis=1)
# Plot distribusi kelas
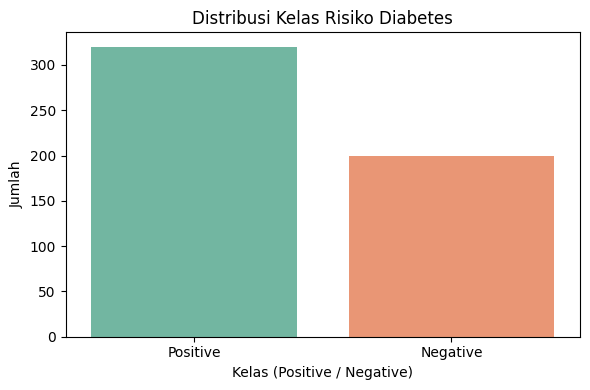
plt.figure(figsize=(6, 4))
sns.countplot(data=data, x='class', palette='Set2')
plt.title('Distribusi Kelas Risiko Diabetes')
plt.xlabel('Kelas (Positive / Negative)')
plt.ylabel('Jumlah')
plt.tight_layout()
plt.show()
/tmp/ipython-input-6-4223954437.py:3: FutureWarning:
Passing `palette` without assigning `hue` is deprecated and will be removed in v0.14.0. Assign the `x` variable to `hue` and set `legend=False` for the same effect.
sns.countplot(data=data, x='class', palette='Set2')
Struktur Dataset#
Fitur dan Tabel#
data.shape #untuk mengetahui bentuk dataset(baris & kolom)
(520, 17)
Terdapat 520 baris dan 17 kolom.
data.columns #untuk mengetahui kolom-kolom yang ada pada dataset
Index(['age', 'gender', 'polyuria', 'polydipsia', 'sudden_weight_loss',
'weakness', 'polyphagia', 'genital_thrush', 'visual_blurring',
'itching', 'irritability', 'delayed_healing', 'partial_paresis',
'muscle_stiffness', 'alopecia', 'obesity', 'class'],
dtype='object')
Fitur adalah atribut atau variabel independen dalam dataset yang digunakan untuk membuat prediksi.
Terdapat 16 fitur pada dataset ini:
age Umur pasien (dalam tahun). Nilai numerik (integer).
gender Jenis kelamin pasien: Male atau Female.
polyuria Sering buang air kecil. Nilai kategorikal: Yes atau No.
polydipsia Rasa haus yang berlebihan. Kategorikal: Yes atau No.
sudden_weight_loss Penurunan berat badan secara tiba-tiba. Kategorikal: Yes atau No.
weakness Merasa lemah atau kurang tenaga. Kategorikal: Yes atau No.
polyphagia Nafsu makan berlebihan. Kategorikal: Yes atau No.
genital_thrush Infeksi jamur pada area genital. Kategorikal: Yes atau No.
visual_blurring Penglihatan kabur. Kategorikal: Yes atau No.
itching Gatal-gatal pada kulit. Kategorikal: Yes atau No.
irritability Mudah marah atau tersinggung. Kategorikal: Yes atau No.
delayed_healing Luka sulit sembuh. Kategorikal: Yes atau No.
partial_paresis Kelemahan otot parsial.Kategorikal: Yes atau No.
muscle_stiffness Kekakuan otot. Kategorikal: Yes atau No.
alopecia Kerontokan rambut. Kategorikal: Yes atau No.
obesity Indikasi pasien mengalami obesitas. Kategorikal: Yes atau No.
Label adalah variabel dependen (target) yang menjadi tujuan dari prediksi model. Dalam dataset ini, label terdapat pada kolom class, yang menunjukkan status risiko diabetes:
Positive: Pasien menunjukkan indikasi risiko diabetes tahap awal.
Negative: Pasien tidak menunjukkan indikasi risiko diabetes.
data.dtypes #untuk mengetahui tipe data dari masing-masing kolom
| 0 | |
|---|---|
| age | int64 |
| gender | object |
| polyuria | object |
| polydipsia | object |
| sudden_weight_loss | object |
| weakness | object |
| polyphagia | object |
| genital_thrush | object |
| visual_blurring | object |
| itching | object |
| irritability | object |
| delayed_healing | object |
| partial_paresis | object |
| muscle_stiffness | object |
| alopecia | object |
| obesity | object |
| class | object |
Identifikasi Kualitas Dataset#
Deteksi Missing Value#
Missing value merupakan data yang hilang pada suatu dataset. Hal ini bisa terjadi oleh beberapa faktor, diantaranya adalah :
Interviewer recording error terjadi akibat kelalaian petugas pengumpul data (pewawancara), misalnya ada sejumlah pertanyaan yang terlewatkan.
Respondent inability error terjadi akibat ketidakmampuan responden dalam memberikan jawaban akurat, misalnya karena tidak memahami pertanyaan, bosan atau kelelahan (respondent fatigue) akhirnya responden mengosongkan sejumlah pertanyaan atau berhenti mengisi kuesioner di tengah jalan.
Unwillingness respondent error terjadi karena responden tidak berkenan memberikan jawaban yang akurat, misalnya pertanyaan soal penghasilan, usia, berat badan, pengalaman melakukan pelanggaran hukum, dll.
Cara penanganan Missing Values :
Mengabaikan dan membuang missing data.
Estimasi parameter.
Imputasi.
Pada kolom di bawah ini akan dilakukan pendeteksian missing values terlebih dahulu pada masing-masing kolom.
# Cek apakah ada missing value di setiap kolom
missing_values = data.isnull().sum()
# Tampilkan hasilnya
print("Jumlah Missing Value per Kolom:\n")
print(missing_values)
# (Opsional) Tampilkan kolom mana saja yang punya missing value
print("\nKolom dengan missing value:")
print(missing_values[missing_values > 0])
Jumlah Missing Value per Kolom:
age 0
gender 0
polyuria 0
polydipsia 0
sudden_weight_loss 0
weakness 0
polyphagia 0
genital_thrush 0
visual_blurring 0
itching 0
irritability 0
delayed_healing 0
partial_paresis 0
muscle_stiffness 0
alopecia 0
obesity 0
class 0
dtype: int64
Kolom dengan missing value:
Series([], dtype: int64)
Didapatkan missing value 0 pada setiap fitur
Prepocessing Data#
Preprocessing dilakukan untuk:
Menghindari bias algoritma akibat skala fitur
Meningkatkan akurasi model
Memastikan data bersih dan siap digunakan
Transformasi Data#
Transformasi data sangat diperlukan dalam preprocessing sebelum digunakan oleh model machine learning, terutama ketika dataset berisi tipe data kategorikal dan numerik yang belum siap diproses langsung oleh algoritma.
Berikut adalah penjelasan mengapa transformasi data diperlukan:
Algoritma ML hanya bisa membaca angka
Standarisasi skala fitur numerik
Akurasi model meningkat
Mencegah error saat training
from sklearn.preprocessing import LabelEncoder
# Salin data agar tidak merusak aslinya
data_encoded = data.copy()
# Loop setiap kolom kategorikal dan encode
for col in data_encoded.columns:
if data_encoded[col].dtype == 'object':
le = LabelEncoder()
data_encoded[col] = le.fit_transform(data_encoded[col])
data_encoded.head()
| age | gender | polyuria | polydipsia | sudden_weight_loss | weakness | polyphagia | genital_thrush | visual_blurring | itching | irritability | delayed_healing | partial_paresis | muscle_stiffness | alopecia | obesity | class | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 40 | 1 | 0 | 1 | 0 | 1 | 0 | 0 | 0 | 1 | 0 | 1 | 0 | 1 | 1 | 1 | 1 |
| 1 | 58 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 1 | 0 | 1 | 0 | 1 |
| 2 | 41 | 1 | 1 | 0 | 0 | 1 | 1 | 0 | 0 | 1 | 0 | 1 | 0 | 1 | 1 | 0 | 1 |
| 3 | 45 | 1 | 0 | 0 | 1 | 1 | 1 | 1 | 0 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 1 |
| 4 | 60 | 1 | 1 | 1 | 1 | 1 | 1 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 |
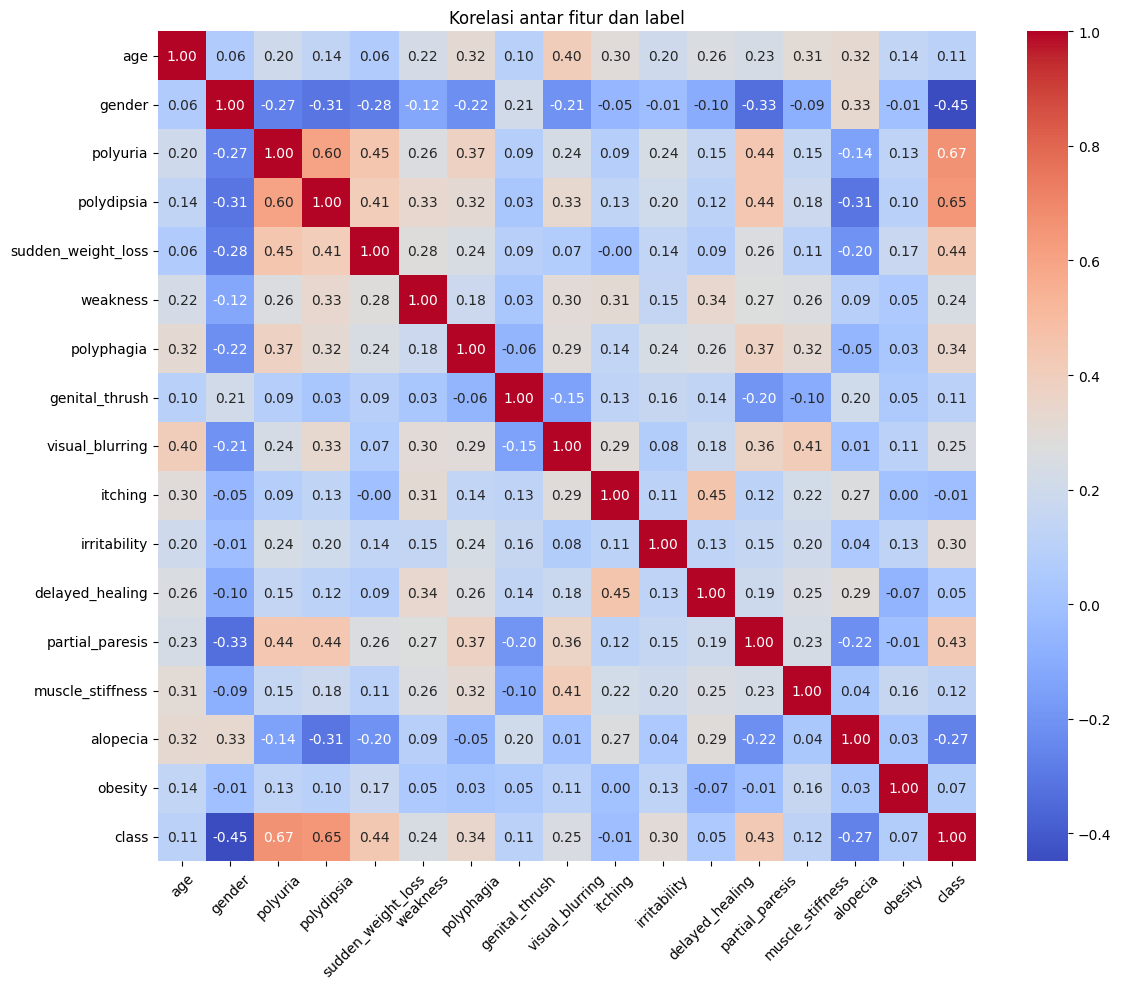
Korelasi antar fitur (setelah encoded)#
Korelasi membantu mengetahui hubungan antar fitur dan dengan target. Jika dua fitur sangat berkorelasi (>0.9), bisa saja salah satu dihapus (redundan). Korelasi dengan label (class) bisa memberi gambaran fitur mana yang paling berpengaruh.
plt.figure(figsize=(12, 10))
sns.heatmap(data_encoded.corr(), annot=True, fmt=".2f", cmap="coolwarm")
plt.title("Korelasi antar fitur dan label")
plt.xticks(rotation=45)
plt.yticks(rotation=0)
plt.tight_layout()
plt.show()
Normalisasi (StandardScaler untuk semua fitur)#
Fitur numerik seperti age memiliki skala besar → ini bisa mengganggu model seperti KNN, SVM, Logistic Regression.
Normalisasi membuat semua fitur setara dalam hal pengaruh terhadap model, dengan rata-rata 0 dan standar deviasi 1.
Tanpa normalisasi, fitur besar bisa dominan secara tidak adil.
from sklearn.preprocessing import StandardScaler
# Pisahkan fitur dan label
X = data_encoded.drop('class', axis=1)
y = data_encoded['class']
# Normalisasi fitur
scaler = StandardScaler()
X_scaled = scaler.fit_transform(X)
# Konversi ke DataFrame agar bisa divisualisasikan
X_scaled_df = pd.DataFrame(X_scaled, columns=X.columns)
X_scaled_df.head()
| age | gender | polyuria | polydipsia | sudden_weight_loss | weakness | polyphagia | genital_thrush | visual_blurring | itching | irritability | delayed_healing | partial_paresis | muscle_stiffness | alopecia | obesity | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | -0.661367 | 0.765092 | -0.992337 | 1.109847 | -0.846269 | 0.839594 | -0.915126 | -0.535844 | -0.901025 | 1.027295 | -0.565506 | 1.084312 | -0.869918 | 1.290994 | 1.380227 | 2.215647 |
| 1 | 0.821362 | 0.765092 | -0.992337 | -0.901025 | -0.846269 | 0.839594 | -0.915126 | -0.535844 | 1.109847 | -0.973430 | -0.565506 | -0.922244 | 1.149534 | -0.774597 | 1.380227 | -0.451335 |
| 2 | -0.578993 | 0.765092 | 1.007722 | -0.901025 | -0.846269 | 0.839594 | 1.092746 | -0.535844 | -0.901025 | 1.027295 | -0.565506 | 1.084312 | -0.869918 | 1.290994 | 1.380227 | -0.451335 |
| 3 | -0.249498 | 0.765092 | -0.992337 | -0.901025 | 1.181657 | 0.839594 | 1.092746 | 1.866215 | -0.901025 | 1.027295 | -0.565506 | 1.084312 | -0.869918 | -0.774597 | -0.724518 | -0.451335 |
| 4 | 0.986110 | 0.765092 | 1.007722 | 1.109847 | 1.181657 | 0.839594 | 1.092746 | -0.535844 | 1.109847 | 1.027295 | 1.768328 | 1.084312 | 1.149534 | 1.290994 | 1.380227 | 2.215647 |
Visualisasi data yang sudah dinormalisasi#
plt.figure(figsize=(12, 5))
sns.boxplot(data=X_scaled_df, orient="h", palette="Set3")
plt.title("Distribusi fitur setelah normalisasi (StandardScaler)")
plt.xlabel("Nilai Standar")
plt.tight_layout()
plt.show()
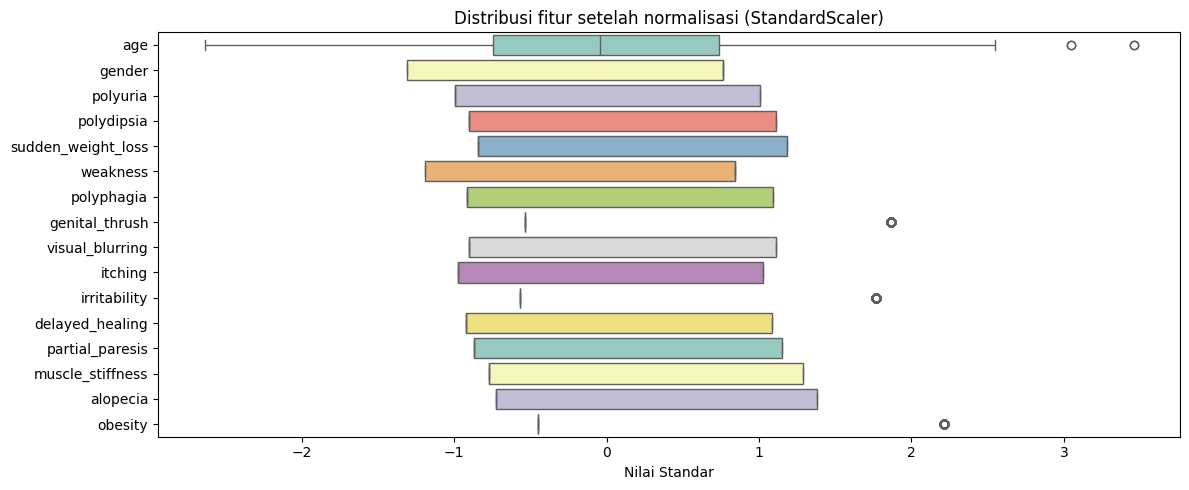
Plot ini menunjukkan bahwa semua fitur sudah berada di skala yang rata-rata mendekati 0, dan terdistribusi seimbang.
Modelling#
Modelling Klasifikasi Gausian Naive Bayes#
Modeling merupakan proses pembuatan dan pengujian model statistik atau matematis yang digunakan untuk menggambarkan dan menganalisis pola atau hubungan dalam data. Tujuan utama dari pemodelan dalam data mining adalah untuk mengidentifikasi pola yang berguna atau prediksi yang akurat dari data yang tersedia.Untuk modelling kali ini bertujuan untuk menentukan class pada suatu data inputan .Data akan dibagi menjadi 2 tipe yaitu data test dan data train dan pemodelan kali ini menggunakan metode Gaussian Naive Bayes.
Pada bagian ini akan ditentukan severity dari data yang akan saya inputan apakah termasuk jinak atau ganas.
cara kalkulasi GNB dapat melalui tahap berikut:
Bagi Dataset menjadi data test dan data train
ada dua jenis pembagian rasio dataset yang sering digunakan yaitu:
80% data train dan 20% data test.
70% data train dan 30% data test.
Untuk kali ini kita akan menggunkan raiso 80% data train dan 20% data test,namun kalian bisa mengubahnya tergantung situasi dan kondisi dilapangan nantinya.
Mengghitung Probabilitas pada data train setiap sheet
lakukan perhitungan probabilitas dari masing-masing kelas sesuai dengan jumlah data train pada kelas tersebut kemudian dibagi dengan banyaknya total data train.
ini digunakan untuk menghitung Prior
Menghitung Mean dan Standart dev
hitung mean dan standart deviasi setiap fitur pada setiap kelas di data train kita
lakukan perhitungan dengan rumus distribusi gaussian dengan rumus sebagai berikut:
hitung posterior
Setelah didapat semua hasil dari distribusi gaussian, langkah selanjutnya adalah menentukan posteriori-nya. Berikut ini untuk rumusnya:
P(A|Hi) : hasil perkalian setiap fitur pada setiap kelas
P(a) : probabilitas setiap kelas
posteriori = P(A|Hi) * P(a)
tentukan maximum posterior
setelah kita menghitung semua posterior maka dapat kita cari mana yang terbesar
data posterior paling besar tersebut yang merupakan akan menjadi class dari data yang kita inputkan
import pandas as pd
from sklearn.naive_bayes import GaussianNB
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import LabelEncoder
from sklearn.metrics import accuracy_score, precision_score, recall_score, f1_score
# Asumsikan kamu sudah punya data asli
# Misal: data = pd.read_csv("early_stage_diabetes_risk_prediction.csv")
# Encode semua kolom kategorikal jadi numerik
data_encoded = data.copy()
for col in data_encoded.columns:
if data_encoded[col].dtype == 'object':
le = LabelEncoder()
data_encoded[col] = le.fit_transform(data_encoded[col])
# Pisahkan fitur dan label
X = data_encoded.drop('class', axis=1)
y = data_encoded['class']
# Split data
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=42)
# Buat model Gaussian Naive Bayes
gnb_model = GaussianNB()
# Latih model
gnb_model.fit(X_train, y_train)
# Prediksi
y_pred = gnb_model.predict(X_test)
# Evaluasi
acc = accuracy_score(y_test, y_pred)
prec = precision_score(y_test, y_pred, average='binary') # binary classification
rec = recall_score(y_test, y_pred, average='binary')
f1 = f1_score(y_test, y_pred, average='binary')
# Tampilkan hasil
print("========== HASIL EVALUASI NAIVE BAYES ==========")
print(f"Akurasi : {acc:.2f}")
print(f"Presisi : {prec:.2f}")
print(f"Recall : {rec:.2f}")
print(f"F1-Score : {f1:.2f}")
# Tampilkan data pelatihan dan pengujian
print("=========================== X TRAINING =================================")
print(X_train)
print("=========================== X TESTING =================================")
print(X_test)
print("=========================== Y TESTING =================================")
print(y_test)
========== HASIL EVALUASI NAIVE BAYES ==========
Akurasi : 0.91
Presisi : 0.93
Recall : 0.94
F1-Score : 0.94
=========================== X TRAINING =================================
age gender polyuria polydipsia sudden_weight_loss weakness \
434 53 1 1 1 1 1
436 41 1 1 1 1 1
208 54 1 0 0 1 1
332 50 0 0 0 0 1
220 38 1 0 0 0 0
.. ... ... ... ... ... ...
71 35 0 0 1 1 1
106 58 1 0 1 1 1
270 40 0 1 1 1 1
435 57 1 1 1 1 1
102 90 0 0 1 1 0
polyphagia genital_thrush visual_blurring itching irritability \
434 1 0 1 0 1
436 1 1 1 1 1
208 0 1 0 0 0
332 0 0 1 1 0
220 0 0 0 0 0
.. ... ... ... ... ...
71 0 0 0 1 0
106 1 0 1 1 0
270 0 0 1 0 0
435 1 0 1 0 0
102 0 1 1 1 0
delayed_healing partial_paresis muscle_stiffness alopecia obesity
434 0 1 0 1 0
436 0 0 0 0 1
208 1 0 0 1 0
332 1 0 0 1 0
220 0 0 0 0 0
.. ... ... ... ... ...
71 1 1 1 0 0
106 0 1 0 1 1
270 1 1 1 0 0
435 0 1 0 0 0
102 0 0 1 1 0
[416 rows x 16 columns]
=========================== X TESTING =================================
age gender polyuria polydipsia sudden_weight_loss weakness \
275 72 1 1 0 0 0
93 40 0 1 1 0 1
6 57 1 1 1 0 1
167 41 1 1 1 1 1
90 45 0 0 0 0 0
.. ... ... ... ... ... ...
69 50 0 1 1 1 1
357 33 0 0 0 0 0
423 66 1 1 1 0 1
334 46 1 0 0 0 1
227 30 1 0 0 1 1
polyphagia genital_thrush visual_blurring itching irritability \
275 1 0 1 1 0
93 1 0 0 1 0
6 1 1 0 0 0
167 1 1 1 1 1
90 1 0 1 1 0
.. ... ... ... ... ...
69 1 0 1 0 0
357 0 0 0 0 0
423 1 0 1 0 0
334 0 0 0 1 0
227 0 0 0 0 0
delayed_healing partial_paresis muscle_stiffness alopecia obesity
275 1 1 1 1 0
93 0 1 0 0 0
6 1 1 0 0 0
167 0 0 0 0 1
90 0 1 0 0 0
.. ... ... ... ... ...
69 0 1 0 0 1
357 0 0 0 0 0
423 0 1 1 0 0
334 1 0 0 1 0
227 0 0 0 0 0
[104 rows x 16 columns]
=========================== Y TESTING =================================
275 0
93 1
6 1
167 1
90 1
..
69 1
357 0
423 1
334 0
227 0
Name: class, Length: 104, dtype: int64
berikut cara menjadikan data train kita ke dalam bentuk csv.
import pandas as pd
# Concatenate X_train and y_train as new column
train_data = pd.concat([X_train, pd.Series(y_train, name='Severity')], axis=1)
# Save training data to CSV with tab-separated values
train_data.to_csv('Train_early_stage_diabetes_risk_prediction.csv', sep="\t", index=False)
uji coba inputan dengan data test menggunakan model yang kita buat.
# Menampilkan data asli baris ke-230 (karena index mulai dari 0)
print("Data yang akan digunakan adalah:")
print(data.iloc[230])
# Siapkan input baru sesuai urutan kolom dataset
# Kolom: ['age', 'gender', 'polyuria', ..., 'obesity']
# Nilai inputan harus sesuai format kategorikal (Yes/No, Male/Female, int)
input_dict = {
'age': 50,
'gender': 'Male',
'polyuria': 'Yes',
'polydipsia': 'Yes',
'sudden_weight_loss': 'No',
'weakness': 'Yes',
'polyphagia': 'No',
'genital_thrush': 'No',
'visual_blurring': 'No',
'itching': 'No',
'irritability': 'No',
'delayed_healing': 'No',
'partial_paresis': 'No',
'muscle_stiffness': 'No',
'alopecia': 'No',
'obesity': 'No'
}
# Ubah input_dict ke DataFrame
input_df = pd.DataFrame([input_dict])
# Encode input sesuai label encoder yang sama
input_encoded = input_df.copy()
for col in input_encoded.columns:
if input_encoded[col].dtype == 'object':
le_input = LabelEncoder()
le_input.fit(data[col]) # gunakan data asli untuk fit encoder
input_encoded[col] = le_input.transform(input_encoded[col])
# Prediksi
prediction = gnb_model.predict(input_encoded)[0]
# Inverse label hasil prediksi
final_result = LabelEncoder()
final_result.fit(data['class'])
predicted_class_label = final_result.inverse_transform([prediction])[0]
# Tampilkan hasil
print("Hasil prediksi:")
print(f"Data yang diinputkan diprediksi sebagai class = {predicted_class_label}")
Data yang akan digunakan adalah:
age 35
gender Male
polyuria No
polydipsia No
sudden_weight_loss Yes
weakness Yes
polyphagia No
genital_thrush Yes
visual_blurring No
itching No
irritability No
delayed_healing No
partial_paresis No
muscle_stiffness No
alopecia No
obesity No
class Negative
Name: 230, dtype: object
Hasil prediksi:
Data yang diinputkan diprediksi sebagai class = Positive
Hasil prediksi: Data yang diinputkan diprediksi sebagai class = Positive
from sklearn.metrics import accuracy_score, precision_score, recall_score, f1_score
# Evaluasi model
accuracy = accuracy_score(y_test, y_pred)
precision = precision_score(y_test, y_pred, average='binary') # karena label hanya 2 kelas
recall = recall_score(y_test, y_pred, average='binary')
fscore = f1_score(y_test, y_pred, average='binary')
# Menampilkan hasil evaluasi
print("========== EVALUASI MODEL NAIVE BAYES ==========")
print(f'Akurasi : {accuracy:.2f}')
print(f'Presisi : {precision:.2f}')
print(f'Recall : {recall:.2f}')
print(f'F1-Score : {fscore:.2f}')
========== EVALUASI MODEL NAIVE BAYES ==========
Akurasi : 0.91
Presisi : 0.93
Recall : 0.94
F1-Score : 0.94
Modelling Klasifikasi KNN#
K-Nearest Neighbors (KNN) merupakan sebuah cara untuk mengklasifikasikan dengan cara melihat sesuatu yang berada di dekatnya. KNN juga disebut dengan Algoritna oembelajar malas. Karena tidak memerlukan pembelajaran terlebih dahulu, dan langsung meyimpan data set dan pada saat klaifikasi melakukan set data. KNN bekerja dengan menggunakan kedekatan dan pemungutan suara mayoritas untuk membuat prediksi atau forecasting. Pada KNN terdapat istilah “k”. “k” merupakan angka yang memberi tahu algoritma beberapa banya titik terdekat (tetangga) yang digunakan untuk membuat keputusan. Contohnya: Misalkan menntukan nama buah dan membandingkannya dengan buah yang telah dikenal. kemudian menggunakan nilai “k” sebanyak 3 kemudian 2 dari 3 merupakan buah apel dan 1 dari 3 merupakan buah pisang. jadi algoritma ini mengatakan bahwa buah tersebut merupakan apel karena sebagian besar tetangganya apel.
Menetukan Nilai “k”#
Dalam KNN, pemilihan nilai “k” sangat penting untuk menentukan hipotesis hasil dari prediksi. Jika kumpulan data memiliki outlier atau noise yang signifikan, nilai “k” yang lebih tinggi dapat membantu memperhalus prediksi dan mengurangi data yang noise. Namun, pemilihan nilai yang tinggi dapat menyebabkan underfitting.
Pertama, kita harus menentukan nilai K terlebih dahulu. Penentuan nilai K ini tidak ada rumus pastinya. Namun satu tips yang dapat dipertimbangkan, yakni jika kelas berjumlah genap maka sebaiknya nilai K-nya ganjil, sebaliknya jika kelas berjumlah ganjil maka sebaiknya nilai K-nya genap. Dalam prakteknya di Python, Anda dapat menghitung menggunakan kode program untuk mencari nilai K terbaik dari berbagai opsi nilai (misalnya dari K=2 sampai K=10).
Hitung jarak antara data baru dan masing-masing data lainnya#
menghitung jarak menggunakan metode Euclidean distance
Jika ada lebih dari satu, kita dapat menjumlahkannya seperti di bawah ini.
Ambil tiga data dengan jarak terdekat. Dari perhitungan Euclidean distance di atas, jika kita rangkum dari jarak terdekat
Impelementasi KNN menggunakan Scikit Learn#
from sklearn.neighbors import KNeighborsClassifier
from sklearn.metrics import accuracy_score, precision_score, recall_score, f1_score, classification_report
from sklearn.preprocessing import LabelEncoder
from sklearn.model_selection import train_test_split
# -------------------- PREPROCESS --------------------
# Salin data dan encode semua kolom kategorikal ke numerik
data_encoded = data.copy()
label_encoders = {} # untuk menyimpan encoder tiap kolom
for col in data_encoded.columns:
if data_encoded[col].dtype == 'object':
le = LabelEncoder()
data_encoded[col] = le.fit_transform(data_encoded[col])
label_encoders[col] = le # simpan untuk nanti
# -------------------- SPLIT --------------------
X = data_encoded.drop('class', axis=1)
y = data_encoded['class'] # sudah dalam bentuk angka (0/1)
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=42)
# -------------------- KNN MODEL --------------------
knn_model = KNeighborsClassifier(n_neighbors=5)
knn_model.fit(X_train, y_train)
y_pred_knn = knn_model.predict(X_test)
# -------------------- EVALUASI --------------------
accuracy_knn = accuracy_score(y_test, y_pred_knn)
precision_knn = precision_score(y_test, y_pred_knn, average='binary')
recall_knn = recall_score(y_test, y_pred_knn, average='binary')
f1_knn = f1_score(y_test, y_pred_knn, average='binary')
# -------------------- OUTPUT --------------------
print("========== EVALUASI MODEL KNN ==========")
print(f'Akurasi : {accuracy_knn:.2f}')
print(f'Presisi : {precision_knn:.2f}')
print(f'Recall : {recall_knn:.2f}')
print(f'F1-Score : {f1_knn:.2f}')
# Tampilkan laporan klasifikasi
print("\n=== Classification Report KNN ===")
class_le = label_encoders['class']
print(classification_report(y_test, y_pred_knn, target_names=class_le.classes_))
========== EVALUASI MODEL KNN ==========
Akurasi : 0.86
Presisi : 0.95
Recall : 0.83
F1-Score : 0.89
=== Classification Report KNN ===
precision recall f1-score support
Negative 0.71 0.91 0.80 33
Positive 0.95 0.83 0.89 71
accuracy 0.86 104
macro avg 0.83 0.87 0.84 104
weighted avg 0.88 0.86 0.86 104
Modelling Klasifikasi Decision Tree#
Decision Tree (Pohon Keputusan) adalah algoritma machine learning yang membangun model prediksi dalam bentuk struktur pohon. Algoritma ini memecah dataset menjadi himpunan data yang lebih kecil dan lebih murni secara bertahap, di mana setiap pemecahan didasarkan pada fitur data yang memberikan informasi paling banyak.
Konsep dan Rumus Inti (Decision Tree - C4.5 / ID3)#
1. Entropy (Ukuran Ketidakpastian)#
Entropy digunakan untuk mengukur tingkat ketidakmurnian atau ketidakpastian dalam dataset. Nilai entropy 0 berarti semua data berada dalam satu kelas, sedangkan nilai lebih tinggi menandakan data tersebar di berbagai kelas.
\(p_i\) = proporsi data pada kelas ke-i dari total data.
2. Information Gain (Pengurangan Ketidakpastian)#
Information Gain digunakan untuk memilih fitur terbaik sebagai node cabang dalam pohon keputusan. Fitur yang memberikan pengurangan ketidakpastian terbesar (gain tertinggi) akan dipilih.
Penerapan pada Dataset Early Stage Diabetes Risk Prediction#
Dataset ini terdiri dari 2 kelas utama:
Positive → Pasien berisiko diabetes
Negative → Pasien tidak berisiko diabetes
Terdapat 16 fitur kategorikal dan numerik, antara lain:
age(numerik)gender,polyuria,polydipsia,sudden_weight_loss,weakness,polyphagia,genital_thrush,visual_blurring,itching,irritability,delayed_healing,partial_paresis,muscle_stiffness,alopecia,obesity(semua kategorikal: Yes/No atau Male/Female)
Karena fitur-fitur pada dataset ini sebagian besar bersifat kategorikal, maka:
Decision Tree akan memisahkan data berdasarkan nilai kategori, seperti:
polyuria = Yes → kanan,No → kiriUntuk fitur numerik seperti
age, decision tree dapat membuat threshold, contoh:
age <= 45.5 → kiri,> 45.5 → kanan
Contoh Proses Pembangunan Pohon#
Langkah 1: Menghitung Entropy Awal (Entropy(S))#
Misalkan:
Jumlah data = 520
Distribusi kelas:
Positive = 320
Negative = 200
Langkah 2: Menghitung Gain untuk Setiap Fitur#
Misal, untuk fitur polyuria, data dipisah berdasarkan nilainya:
\(S_1\): data dengan
polyuria = Yes\(S_2\): data dengan
polyuria = No
Hitung entropy masing-masing subset, lalu gunakan rumus:
Lakukan hal serupa untuk fitur lain. Fitur dengan nilai gain tertinggi akan dijadikan node cabang.
Contoh Struktur Pohon Hasil Training (Misal)#
polyuria = Yes?
|
|--- Yes ---> polydipsia = Yes?
| |
| |--- Yes ---> class: Positive
| |--- No ----> class: Positive
|
|--- No --> itching = Yes?
|
|--- Yes ---> class: Negative
|--- No ----> class: Negative
Implementasi Decision Tree dengan Scikit-learn#
etelah kita memahami cara kerja Decision Tree secara manual dengan menghitung Entropy dan Information Gain, kini saatnya kita melihat bagaimana proses ini dilakukan secara otomatis menggunakan library machine learning populer, yaitu Scikit-learn (sklearn). Scikit-learn menyediakan implementasi Decision Tree yang efisien dan siap pakai.
Kita akan menggunakan data yang sudah didiskretisasi sebelumnya, lalu melatih model DecisionTreeClassifier dari Scikit-learn untuk membangun pohon keputusan. Hasil pohon yang dibangun oleh Scikit-learn ini seharusnya konsisten dengan logika perhitungan manual yang sudah kita lakukan.
Terakhir, kita akan memvisualisasikan pohon yang dihasilkan oleh Scikit-learn untuk melihat strukturnya secara grafis.
import pandas as pd
from ucimlrepo import fetch_ucirepo
from sklearn.preprocessing import LabelEncoder
from sklearn.model_selection import train_test_split
from sklearn.tree import DecisionTreeClassifier, plot_tree
from sklearn.metrics import accuracy_score, precision_score, recall_score, f1_score, classification_report
import matplotlib.pyplot as plt
import joblib
# Fetch dataset dari UCI
diabetes_data = fetch_ucirepo(id=529)
# Ambil fitur dan label
X = diabetes_data.data.features
y = diabetes_data.data.targets
# Gabungkan jika ingin menyimpan atau melihat struktur lengkap
data = pd.concat([X, y], axis=1)
# Ubah seluruh kolom kategorikal menjadi numerik dengan LabelEncoder
X_encoded = X.copy()
for col in X_encoded.columns:
if X_encoded[col].dtype == 'object':
X_encoded[col] = LabelEncoder().fit_transform(X_encoded[col])
# Encode label target ('Positive'/'Negative')
le = LabelEncoder()
y_encoded = le.fit_transform(y)
# Split train-test
X_train, X_test, y_train, y_test = train_test_split(X_encoded, y_encoded, test_size=0.2, random_state=0)
# Simpan model Decision Tree
joblib.dump(dt_model, 'decision_tree_model.pkl')
/usr/local/lib/python3.11/dist-packages/sklearn/preprocessing/_label.py:110: DataConversionWarning: A column-vector y was passed when a 1d array was expected. Please change the shape of y to (n_samples, ), for example using ravel().
y = column_or_1d(y, warn=True)
['decision_tree_model.pkl']
Training Decison Tree#
# Buat dan latih model Decision Tree dengan entropy
dt_model = DecisionTreeClassifier(criterion='entropy', max_depth=4, random_state=0)
dt_model.fit(X_train, y_train)
# Prediksi
y_pred_dt = dt_model.predict(X_test)
Evaluasi Model Decision Tree#
# Evaluasi kinerja
accuracy_dt = accuracy_score(y_test, y_pred_dt)
precision_dt = precision_score(y_test, y_pred_dt, average='macro')
recall_dt = recall_score(y_test, y_pred_dt, average='macro')
f1_dt = f1_score(y_test, y_pred_dt, average='macro')
# Tampilkan hasil evaluasi
print("========== EVALUASI MODEL DECISION TREE ==========")
print(f'Akurasi : {accuracy_dt:.2f}')
print(f'Presisi : {precision_dt:.2f}')
print(f'Recall : {recall_dt:.2f}')
print(f'F1-Score: {f1_dt:.2f}')
# Tampilkan klasifikasi per kelas
print("\n=== Classification Report ===")
print(classification_report(y_test, y_pred_dt, target_names=le.classes_))
========== EVALUASI MODEL DECISION TREE ==========
Akurasi : 0.94
Presisi : 0.94
Recall : 0.94
F1-Score: 0.94
=== Classification Report ===
precision recall f1-score support
Negative 0.93 0.93 0.93 40
Positive 0.95 0.95 0.95 64
accuracy 0.94 104
macro avg 0.94 0.94 0.94 104
weighted avg 0.94 0.94 0.94 104
Visualisasi Pohon Keputusan#
# Visualisasi pohon keputusan
plt.figure(figsize=(22,10))
plot_tree(dt_model,
feature_names=X_encoded.columns, # kolom numerik hasil LabelEncoder
class_names=le.classes_, # nama label asli (Positive/Negative)
filled=True,
rounded=True)
plt.title("Visualisasi Pohon Keputusan (Decision Tree untuk Prediksi Diabetes)")
plt.show()
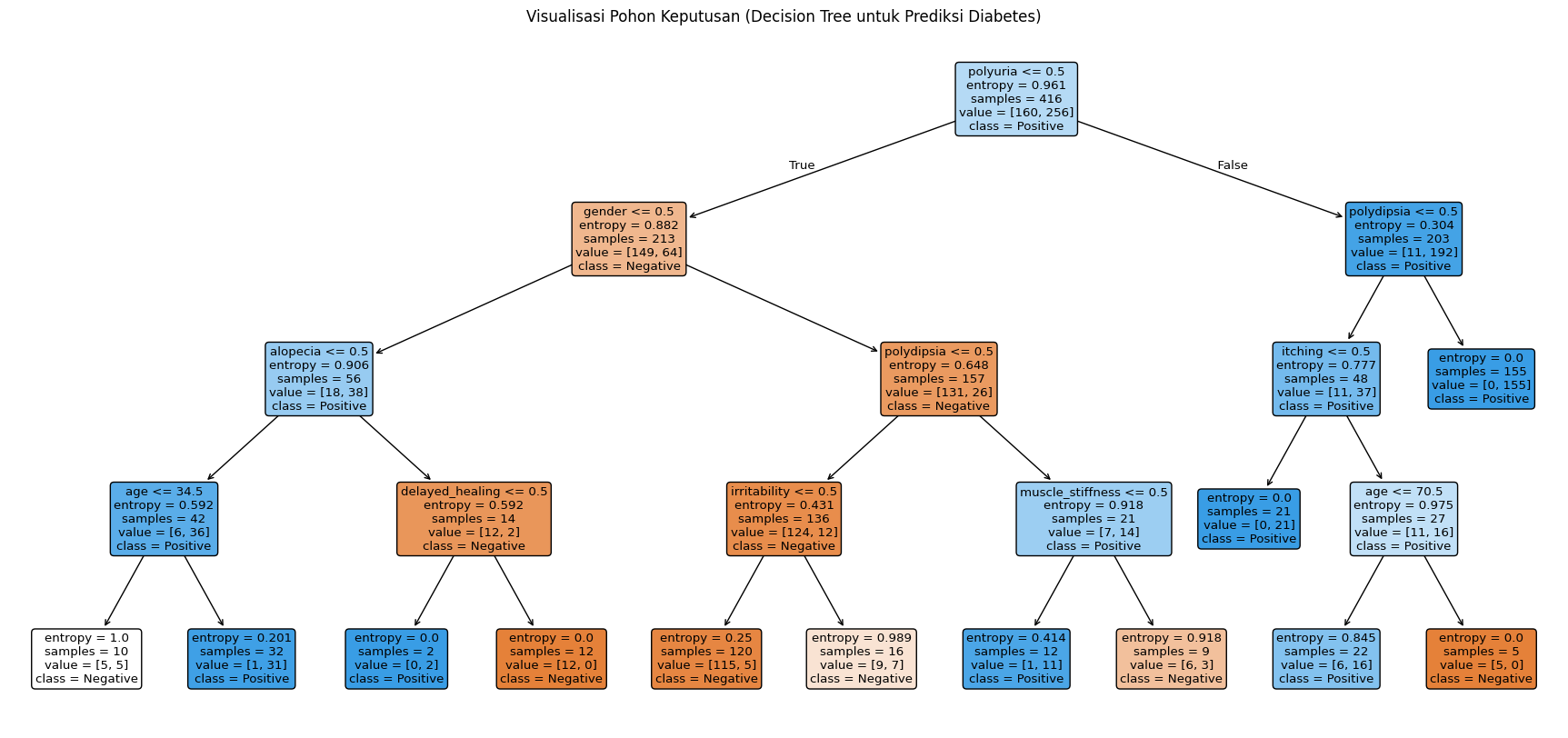
Pohon keputusan yang dihasilkan menggunakan algoritma C4.5 / ID3 dengan criterion=’entropy’ menunjukkan struktur pembelajaran berdasarkan fitur yang paling berpengaruh terhadap klasifikasi diabetes positif atau negatif.
🌳 Node Akar (Root Node):
Fitur paling berpengaruh pertama: polyuria
Threshold awal: polyuria <= 0.5 Artinya, pasien yang tidak mengalami polyuria lebih cenderung Negatif, sementara pasien yang mengalami polyuria lebih cenderung Positif.
Berdasarkan urutan pembelahan pada pohon:
polyuria: Gejala sering buang air kecil – menjadi fitur paling krusial.
polydipsia: Rasa haus berlebihan – muncul pada cabang kanan dan kiri.
gender, alopecia, itching, irritability, age, dan delayed_healing juga berkontribusi, terutama untuk klasifikasi lebih lanjut.
Evaluasi#
Dari ketiga model yang dibuat dapat dilihat akurasi dari masing-masing model, yaitu:
Naive Bayes: memiliki akurasi 0.91
KNN: memiliki akurasi 0.86
Decision Tree: memiliki akurasi 0.94
Dari ketiga model diatas dapat disimpulkan bahwa single model Decision Tree memiliki akurasi tertinggi yaitu 94%.
Deployment#
Menyimpan Model#
pada tahap ini saya akan menyimpan model dari single model Decision Tree yang sudah dibuat. Tujuan dilakukan penyimpanan model adalah supaya mudah dipanggil ketika akan memasuki tahap deployment.
Untuk library yang saya gunakan dalam menyimpan model yaitu joblib.
import joblib
# Simpan model Decision Tree
joblib.dump(dt_model, 'decision_tree_model.pkl')
# Load kembali model Decision Tree
dt_model = joblib.load('decision_tree_model.pkl')
joblib.dump(le, 'label_encoder.pkl')
['label_encoder.pkl']
from google.colab import files
files.download('decision_tree_model.pkl')
from google.colab import files
files.download('label_encoder.pkl')
Integrasi Model Dengan Web#
install library Streamlit, dengan menjalankan perintah “pip install streamlit” pada terminal.
membuat file berekstensi .py, misal app.py.
didalam app.py saya panggil model yang telah disimpan.
setelah itu saya membuat tampilan informasi keterangan tentang inputan dan hasil evaluasi. selain itu saya juga membuat tampilan form untuk user menginputkan data yang akan diprediksi oleh model berserta tampilan untuk hasil prediksi. untuk tampilan saya masukkan code nya didalam file app.py juga, karena dimudahkan oleh library streamlit.
Source code yang telah saya buat dapat diakses melalui link berikut :
Hasil Web#
link web: https://uaspendatayudhacaesarmaulana.streamlit.app/
Berikut adalah hasil dari webnya. Terdapat form untuk memasukkan fitur-fitur yang nantinya akan diprediksi.
