Prediksi Tulang Belakang Normal, Disk Hernia, atau Spondylolisthesis#
Pendahuluan#
Permasalahan kelainan tulang belakang merupakan salah satu gangguan kesehatan yang sering terjadi pada masyarakat, terutama akibat faktor usia, postur tubuh yang buruk, maupun beban kerja fisik yang berlebihan. Dua jenis kelainan yang umum ditemukan adalah disk hernia dan spondylolisthesis, yang dapat menyebabkan rasa nyeri, keterbatasan gerak, dan penurunan kualitas hidup. Deteksi dini terhadap kelainan-kelainan tersebut sangat penting agar penanganan medis dapat dilakukan secepat mungkin.
Dengan berkembangnya teknologi, khususnya di bidang kecerdasan buatan. proses klasifikasi atau diagnosis kelainan tulang belakang kini dapat dibantu oleh sistem cerdas berbasis data.
Data Understanding#
Sumber Data#
Dataset diambil dari link dibawah ini:
https://archive.ics.uci.edu/dataset/212/vertebral+column
Salah satu dataset yang umum digunakan dalam pengembangan dan penelitian klasifikasi medis adalah Vertebral Column Dataset yang tersedia di UCI Machine Learning Repository. Dataset ini memuat parameter biomekanis yang diambil dari citra radiografi tulang belakang pasien, seperti pelvic incidence, sacral slope, dan lumbar lordosis angle.
Tujuan#
Penelitian ini bertujuan untuk mengimplementasikan algoritma klasifikasi dalam mengelompokkan kondisi tulang belakang pasien ke dalam tiga kategori, yaitu: normal, hernia diskus, dan spondylolisthesis. Dengan memanfaatkan teknik pembelajaran mesin, diharapkan sistem ini mampu menjadi alat bantu diagnosis awal yang efisien, akurat, dan bermanfaat dalam bidang medis, khususnya ortopedi.
Integrasi Data#
untuk mengambil data agar dapat diolah, perlu untuk menginstall package yang telah disediakan oleh UCI Dataset. Instalasi dilakukan berguna untuk menarik data yang berasal dari UCI dataset agar dapat diolah. peritah untuk mengambil data dari UCI dataset dapat di lihat ketika menekan tombol import in python pada datase yang diinginkan dan ikuti perintah tersebut agar data dapat diambil dari UCI dataset. Contoh pengambilan data dari UCI dataset dapat dilihat pada gambar dan perintah berikut:
!pip install ucimlrepo
Requirement already satisfied: ucimlrepo in /usr/local/python/3.12.1/lib/python3.12/site-packages (0.0.7)
Requirement already satisfied: pandas>=1.0.0 in /home/codespace/.local/lib/python3.12/site-packages (from ucimlrepo) (2.2.3)
Requirement already satisfied: certifi>=2020.12.5 in /home/codespace/.local/lib/python3.12/site-packages (from ucimlrepo) (2024.8.30)
Requirement already satisfied: numpy>=1.26.0 in /home/codespace/.local/lib/python3.12/site-packages (from pandas>=1.0.0->ucimlrepo) (2.2.0)
Requirement already satisfied: python-dateutil>=2.8.2 in /home/codespace/.local/lib/python3.12/site-packages (from pandas>=1.0.0->ucimlrepo) (2.9.0.post0)
Requirement already satisfied: pytz>=2020.1 in /home/codespace/.local/lib/python3.12/site-packages (from pandas>=1.0.0->ucimlrepo) (2024.2)
Requirement already satisfied: tzdata>=2022.7 in /home/codespace/.local/lib/python3.12/site-packages (from pandas>=1.0.0->ucimlrepo) (2024.2)
Requirement already satisfied: six>=1.5 in /home/codespace/.local/lib/python3.12/site-packages (from python-dateutil>=2.8.2->pandas>=1.0.0->ucimlrepo) (1.17.0)
[notice] A new release of pip is available: 24.3.1 -> 25.1.1
[notice] To update, run: python3 -m pip install --upgrade pip
from ucimlrepo import fetch_ucirepo
# fetch dataset
vertebral_column = fetch_ucirepo(id=212)
# data (as pandas dataframes)
X = vertebral_column.data.features
y = vertebral_column.data.targets
data = vertebral_column.data.original
data.to_csv("vertebral_column.csv", index=True)
print(data.info()) #untuk menampilkan info fitur-fitur yang ada di tabel
print(data.head()) #untuk menampilkan 5 baris pertama
<class 'pandas.core.frame.DataFrame'>
RangeIndex: 310 entries, 0 to 309
Data columns (total 7 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 pelvic_incidence 310 non-null float64
1 pelvic_tilt 310 non-null float64
2 lumbar_lordosis_angle 310 non-null float64
3 sacral_slope 310 non-null float64
4 pelvic_radius 310 non-null float64
5 degree_spondylolisthesis 310 non-null float64
6 class 310 non-null object
dtypes: float64(6), object(1)
memory usage: 17.1+ KB
None
pelvic_incidence pelvic_tilt lumbar_lordosis_angle sacral_slope \
0 63.027817 22.552586 39.609117 40.475232
1 39.056951 10.060991 25.015378 28.995960
2 68.832021 22.218482 50.092194 46.613539
3 69.297008 24.652878 44.311238 44.644130
4 49.712859 9.652075 28.317406 40.060784
pelvic_radius degree_spondylolisthesis class
0 98.672917 -0.254400 Hernia
1 114.405425 4.564259 Hernia
2 105.985135 -3.530317 Hernia
3 101.868495 11.211523 Hernia
4 108.168725 7.918501 Hernia
Eksplorasi Data#
VIsualisasi Data#
display(data) #display dataset
| pelvic_incidence | pelvic_tilt | lumbar_lordosis_angle | sacral_slope | pelvic_radius | degree_spondylolisthesis | class | |
|---|---|---|---|---|---|---|---|
| 0 | 63.027817 | 22.552586 | 39.609117 | 40.475232 | 98.672917 | -0.254400 | Hernia |
| 1 | 39.056951 | 10.060991 | 25.015378 | 28.995960 | 114.405425 | 4.564259 | Hernia |
| 2 | 68.832021 | 22.218482 | 50.092194 | 46.613539 | 105.985135 | -3.530317 | Hernia |
| 3 | 69.297008 | 24.652878 | 44.311238 | 44.644130 | 101.868495 | 11.211523 | Hernia |
| 4 | 49.712859 | 9.652075 | 28.317406 | 40.060784 | 108.168725 | 7.918501 | Hernia |
| ... | ... | ... | ... | ... | ... | ... | ... |
| 305 | 47.903565 | 13.616688 | 36.000000 | 34.286877 | 117.449062 | -4.245395 | Normal |
| 306 | 53.936748 | 20.721496 | 29.220534 | 33.215251 | 114.365845 | -0.421010 | Normal |
| 307 | 61.446597 | 22.694968 | 46.170347 | 38.751628 | 125.670725 | -2.707880 | Normal |
| 308 | 45.252792 | 8.693157 | 41.583126 | 36.559635 | 118.545842 | 0.214750 | Normal |
| 309 | 33.841641 | 5.073991 | 36.641233 | 28.767649 | 123.945244 | -0.199249 | Normal |
310 rows × 7 columns
Tampilkan banyaknya class#
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
from ucimlrepo import fetch_ucirepo
# Ambil dataset
vertebral_column = fetch_ucirepo(id=212)
# Gabungkan fitur dan target
X = vertebral_column.data.features
y = vertebral_column.data.targets
data = pd.concat([X, y], axis=1)
plt.figure(figsize=(6, 4))
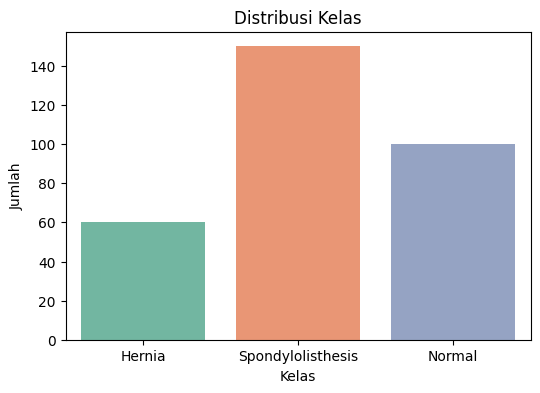
sns.countplot(data=data, x='class', palette='Set2')
plt.title('Distribusi Kelas')
plt.xlabel('Kelas')
plt.ylabel('Jumlah')
plt.show()
/tmp/ipykernel_2773/468467621.py:2: FutureWarning:
Passing `palette` without assigning `hue` is deprecated and will be removed in v0.14.0. Assign the `x` variable to `hue` and set `legend=False` for the same effect.
sns.countplot(data=data, x='class', palette='Set2')
Struktur Dataset#
Fitur dan Tabel#
data.shape #untuk mengetahui bentuk dataset(baris & kolom)
(310, 7)
Terdapat 310 baris dan 7 kolom.
data.columns #untuk mengetahui kolom-kolom yang ada pada dataset
Index(['pelvic_incidence', 'pelvic_tilt', 'lumbar_lordosis_angle',
'sacral_slope', 'pelvic_radius', 'degree_spondylolisthesis', 'class'],
dtype='object')
Fitur adalah atribut atau variabel independen dalam dataset yang digunakan untuk membuat prediksi.
Terdapat 6 fitur pada dataset ini:
pelvic_incidence: Sudut antara garis tegak lurus terhadap sakrum dan garis yang menghubungkan pusat kepala femur. (Nilai numerik dalam derajat (°))
pelvic_tilt: Mengukur kemiringan panggul terhadap garis vertikal. Ini mencerminkan posisi panggul terhadap postur tubuh. (Nilai derajat kemiringa)
lumbar_lordosis_angle: udut kelengkungan tulang belakang bagian bawah (lumbar). (Nilai numerik dalam derajat)
sacral_slope: Sudut kemiringan sakrum terhadap horizontal. Ini berbanding langsung dengan pelvic incidence. (Nilai numerik deraja)
pelvic_radius: Jarak antara pusat sakrum dan kepala femur. Merupakan parameter ukuran anatomi panggul. (Nilai panjang (kemungkinan satuannya mm atau cm))
degree_spondylolisthesis: Derajat pergeseran atau “slip” tulang belakang ke depan terhadap ruas bawahnya. (Nilai real/float yang bisa positif atau negatif)
Label adalah variabel dependen atau target yang ingin diprediksi oleh model machine learning. Label merupakan output yang dipelajari oleh model dari data.
Terdapat 1 field label dengan 3 tipe pada dataset ini:
Normal
Disk Hernia
spondylolisthesis
data.dtypes #untuk mengetahui tipe data dari masing-masing kolom
pelvic_incidence float64
pelvic_tilt float64
lumbar_lordosis_angle float64
sacral_slope float64
pelvic_radius float64
degree_spondylolisthesis float64
class object
dtype: object
Identifikasi Kualitas Dataset#
Deteksi Missing Value#
Missing value merupakan data yang hilang pada suatu dataset. Hal ini bisa terjadi oleh beberapa faktor, diantaranya adalah :
Interviewer recording error terjadi akibat kelalaian petugas pengumpul data (pewawancara), misalnya ada sejumlah pertanyaan yang terlewatkan.
Respondent inability error terjadi akibat ketidakmampuan responden dalam memberikan jawaban akurat, misalnya karena tidak memahami pertanyaan, bosan atau kelelahan (respondent fatigue) akhirnya responden mengosongkan sejumlah pertanyaan atau berhenti mengisi kuesioner di tengah jalan.
Unwillingness respondent error terjadi karena responden tidak berkenan memberikan jawaban yang akurat, misalnya pertanyaan soal penghasilan, usia, berat badan, pengalaman melakukan pelanggaran hukum, dll.
Cara penanganan Missing Values :
Mengabaikan dan membuang missing data.
Estimasi parameter.
Imputasi.
pada kolom di bawah ini akan dilakukan pendeteksian missing values terlebih dahulu pada masing-masing kolom.
# Cek apakah ada missing value di setiap kolom
missing_values = data.isnull().sum()
# Tampilkan hasilnya
print("Jumlah Missing Value per Kolom:\n")
print(missing_values)
# (Opsional) Tampilkan kolom mana saja yang punya missing value
print("\nKolom dengan missing value:")
print(missing_values[missing_values > 0])
Jumlah Missing Value per Kolom:
pelvic_incidence 0
pelvic_tilt 0
lumbar_lordosis_angle 0
sacral_slope 0
pelvic_radius 0
degree_spondylolisthesis 0
class 0
dtype: int64
Kolom dengan missing value:
Series([], dtype: int64)
Didapatkan missing value 0 pada setiap fitur
Prepocessing Data#
Preprocessing dilakukan untuk:
Menghindari bias algoritma akibat skala fitur
Meningkatkan akurasi model
Memastikan data bersih dan siap digunakan
Normalisasi#
Karena penelitian ini akan menggunakan KNN maka perlu normalisasi karean algoritma ini sensitif terhadap jarak antar fitur
Naive Bayes dan Decision Tree sebenarnya tidak terlalu bergantung pada skala, namun tetap direkomendasikan untuk menjaga konsistensi.
from sklearn.preprocessing import StandardScaler
# Normalisasi fitur (tanpa label)
scaler = StandardScaler()
X_scaled = scaler.fit_transform(X)
# Konversi kembali ke DataFrame (jika ingin melihat)
X_scaled_df = pd.DataFrame(X_scaled, columns=X.columns)
X_scaled_df.head()
| pelvic_incidence | pelvic_tilt | lumbar_lordosis_angle | sacral_slope | pelvic_radius | degree_spondylolisthesis | |
|---|---|---|---|---|---|---|
| 0 | 0.147086 | 0.501369 | -0.665177 | -0.184950 | -1.447647 | -0.708059 |
| 1 | -1.245864 | -0.748769 | -1.453001 | -1.041521 | -0.264385 | -0.579556 |
| 2 | 0.484370 | 0.467932 | -0.099262 | 0.273083 | -0.897686 | -0.795421 |
| 3 | 0.511390 | 0.711562 | -0.411339 | 0.126128 | -1.207303 | -0.402288 |
| 4 | -0.626648 | -0.789693 | -1.274745 | -0.215876 | -0.733455 | -0.490106 |
Deteksi dan Penanganan Outlier#
Outlier/pencilan merupakan data pada dataset yang menyimpang dari data lainnya,mendeteksi outlier perlu agar data yang diolah memberikan hasil yang baik pada model yang akan dibuat nantinya. Outlier dapat diidentifikasi dan ditangani dengan beberapa cara antara lain yang akan kami gunakan yaitu LOF(Local Outlier Factor).
Konsep Local Outlier Factor#
Outlier adalah titik data yang berbeda atau jauh dari titik data lainnya. Local Outlier Factor (LOF) adalah algoritma yang mengidentifikasi outlier yang ada dalam kumpulan data. Ketika suatu titik dianggap sebagai outlier berdasarkan lingkungan lokalnya, maka titik tersebut disebut local outlier. LOF akan mengidentifikasi outlier dengan mempertimbangkan kepadatan lingkungan. LOF bekerja dengan baik ketika kepadatan data tidak sama di seluruh kumpulan data.
Untuk memahami LOF, ada beberapa konsep yang harus dipelajari secara berurutan:
K-distance dan K-neighbors
Reachability Distance (RD)
Local Reachability Density (LRD)
Local Outlier Factor (LOF)
K-distance & K-neighbors
K-distance adalah jarak antara suatu titik, dan merupakan tetangga terdekat Kᵗʰ. Tetangga K yang dilambangkan dengan Nₖ(A) mencakup himpunan titik yang terletak di dalam atau pada lingkaran berjari-jari jarak K. K-tetangga bisa lebih dari atau sama dengan nilai K. Kita akan melihat contohnya. Katakanlah kita mempunyai empat titik A, B, C, dan D. Jika K=2, K-tetangga A adalah C, B, dan D. Di sini, nilai K=2 tetapi ||N₂(A)|| = 3. Oleh karena itu, ||Nₖ(titik)|| akan selalu lebih besar atau sama dengan K.
Reachability Distance (RD)
didefinisikan sebagai jarak K maksimum Xj dan jarak antara Xi dan Xj. Ukuran jarak bersifat khusus untuk masalah (Euclidean, Manhattan, dll.) Dalam istilah awam, jika titik Xi terletak di dalam K-tetangga Xj, maka jarak jangkauannya adalah K-jarak Xj (garis biru), jika tidak, jarak jangkauannya adalah jarak antara Xi dan Xj (garis oranye).
Local reachability density (LRD)
LRD merupakan kebalikan dari rata-rata jarak jangkauan A dari tetangganya. Semakin besar jarak jangkauan rata-rata (yaitu, tetangga jauh dari titik tersebut), semakin sedikit kepadatan titik yang ada di sekitar titik tertentu. Ini menunjukkan seberapa jauh suatu titik dari kelompok titik terdekat. Nilai LRD yang rendah menunjukkan bahwa cluster terdekat berada jauh dari titik.
Local Outlier Factor (LOF)
LRD tiap titik digunakan untuk membandingkan dengan rata-rata LRD K tetangganya. LOF adalah perbandingan rata-rata LRD K tetangga A terhadap LRD A. Jika suatu titik bukan merupakan pencilan (inlier), rasio rata-rata LRD tetangganya kira-kira sama dengan LRD suatu titik (karena kepadatan suatu titik dan tetangganya kira-kira sama). Dalam hal ini, LOF hampir sama dengan 1. Sebaliknya, jika suatu titik merupakan outlier, LRD suatu titik lebih kecil dari rata-rata LRD tetangganya. Maka nilai LOF akan tinggi. Umumnya jika LOF > 1 maka dianggap outlier, namun hal tersebut tidak selalu benar. Katakanlah kita mengetahui bahwa kita hanya memiliki satu outlier dalam data, lalu kita ambil nilai LOF maksimum di antara semua nilai LOF, dan titik yang sesuai dengan nilai LOF maksimum akan dianggap sebagai outlier.
Untuk langkah-langkah perhitungan manual Local Outlier Factor (LOF) yaitu :
Menghitung jarak dan menentukan tetangga
Menghitung Reachability Distance (RD)
Menghitung Local Reachability Distance (LRD)
Menghitung Local Outlier Factor (LOF)
Implementasi Local Outlier Factor dengan Scikit Learn#
from sklearn.neighbors import LocalOutlierFactor
import matplotlib.pyplot as plt
import pandas as pd
# Pastikan data tersedia dan label terakhir dihapus
data1 = data.drop(columns=['class']) # Buang label untuk analisis outlier
# Membuat model LOF
lof = LocalOutlierFactor(n_neighbors=9, contamination=0.03)
# Menyimpan indeks outlier dalam list
outlier_indices = []
# Melakukan deteksi outlier untuk setiap fitur secara terpisah
for column in data1.columns:
feature_values = data1[column].values.reshape(-1, 1)
y_pred = lof.fit_predict(feature_values)
# Tambahkan indeks yang dideteksi sebagai outlier (label -1)
outlier_indices.extend([(i, column) for i, label in enumerate(y_pred) if label == -1])
# Menghilangkan duplikat indeks
outlier_indices = list(set(outlier_indices))
# Menampilkan informasi outlier
print("===================================== OUTLIER =====================================")
for i in outlier_indices:
print(f"Outlier ditemukan pada baris => {i[0]}, kolom => {i[1]}")
# Menyusun indeks baris yang mengandung outlier (tanpa duplikat)
outlier_row_indices = sorted(set([i[0] for i in outlier_indices]))
# Tampilkan baris yang mengandung outlier
outlier_data = data.iloc[outlier_row_indices]
print("===================================== DATA =====================================")
print("Data pada baris yang mengandung outlier:")
print(outlier_data)
===================================== OUTLIER =====================================
Outlier ditemukan pada baris => 39, kolom => pelvic_incidence
Outlier ditemukan pada baris => 115, kolom => degree_spondylolisthesis
Outlier ditemukan pada baris => 7, kolom => degree_spondylolisthesis
Outlier ditemukan pada baris => 179, kolom => pelvic_tilt
Outlier ditemukan pada baris => 95, kolom => degree_spondylolisthesis
Outlier ditemukan pada baris => 166, kolom => pelvic_radius
Outlier ditemukan pada baris => 11, kolom => lumbar_lordosis_angle
Outlier ditemukan pada baris => 94, kolom => pelvic_incidence
Outlier ditemukan pada baris => 162, kolom => pelvic_incidence
Outlier ditemukan pada baris => 206, kolom => pelvic_incidence
Outlier ditemukan pada baris => 11, kolom => sacral_slope
Outlier ditemukan pada baris => 163, kolom => sacral_slope
Outlier ditemukan pada baris => 141, kolom => pelvic_tilt
Outlier ditemukan pada baris => 96, kolom => pelvic_incidence
Outlier ditemukan pada baris => 26, kolom => lumbar_lordosis_angle
Outlier ditemukan pada baris => 24, kolom => sacral_slope
Outlier ditemukan pada baris => 290, kolom => lumbar_lordosis_angle
Outlier ditemukan pada baris => 26, kolom => sacral_slope
Outlier ditemukan pada baris => 75, kolom => degree_spondylolisthesis
Outlier ditemukan pada baris => 192, kolom => pelvic_radius
Outlier ditemukan pada baris => 37, kolom => lumbar_lordosis_angle
Outlier ditemukan pada baris => 26, kolom => degree_spondylolisthesis
Outlier ditemukan pada baris => 235, kolom => degree_spondylolisthesis
Outlier ditemukan pada baris => 112, kolom => pelvic_tilt
Outlier ditemukan pada baris => 248, kolom => lumbar_lordosis_angle
Outlier ditemukan pada baris => 168, kolom => pelvic_incidence
Outlier ditemukan pada baris => 257, kolom => pelvic_tilt
Outlier ditemukan pada baris => 37, kolom => sacral_slope
Outlier ditemukan pada baris => 304, kolom => pelvic_radius
Outlier ditemukan pada baris => 147, kolom => pelvic_tilt
Outlier ditemukan pada baris => 94, kolom => sacral_slope
Outlier ditemukan pada baris => 162, kolom => sacral_slope
Outlier ditemukan pada baris => 197, kolom => lumbar_lordosis_angle
Outlier ditemukan pada baris => 96, kolom => sacral_slope
Outlier ditemukan pada baris => 228, kolom => degree_spondylolisthesis
Outlier ditemukan pada baris => 75, kolom => pelvic_radius
Outlier ditemukan pada baris => 10, kolom => degree_spondylolisthesis
Outlier ditemukan pada baris => 115, kolom => pelvic_incidence
Outlier ditemukan pada baris => 142, kolom => lumbar_lordosis_angle
Outlier ditemukan pada baris => 167, kolom => pelvic_radius
Outlier ditemukan pada baris => 151, kolom => pelvic_tilt
Outlier ditemukan pada baris => 287, kolom => lumbar_lordosis_angle
Outlier ditemukan pada baris => 206, kolom => pelvic_tilt
Outlier ditemukan pada baris => 252, kolom => pelvic_tilt
Outlier ditemukan pada baris => 147, kolom => pelvic_radius
Outlier ditemukan pada baris => 168, kolom => sacral_slope
Outlier ditemukan pada baris => 122, kolom => pelvic_tilt
Outlier ditemukan pada baris => 54, kolom => degree_spondylolisthesis
Outlier ditemukan pada baris => 83, kolom => pelvic_radius
Outlier ditemukan pada baris => 180, kolom => pelvic_radius
Outlier ditemukan pada baris => 278, kolom => degree_spondylolisthesis
Outlier ditemukan pada baris => 163, kolom => pelvic_incidence
Outlier ditemukan pada baris => 263, kolom => pelvic_tilt
Outlier ditemukan pada baris => 49, kolom => lumbar_lordosis_angle
Outlier ditemukan pada baris => 85, kolom => pelvic_radius
Outlier ditemukan pada baris => 189, kolom => pelvic_incidence
Outlier ditemukan pada baris => 173, kolom => pelvic_radius
Outlier ditemukan pada baris => 40, kolom => lumbar_lordosis_angle
Outlier ditemukan pada baris => 115, kolom => sacral_slope
Outlier ditemukan pada baris => 26, kolom => pelvic_incidence
===================================== DATA =====================================
Data pada baris yang mengandung outlier:
pelvic_incidence pelvic_tilt lumbar_lordosis_angle sacral_slope \
7 45.366754 10.755611 29.038349 34.611142
10 49.706610 13.040974 31.334500 36.665635
11 31.232387 17.715819 15.500000 13.516568
24 36.125683 22.758753 29.000000 13.366931
26 26.147921 10.759454 14.000000 15.388468
37 35.703458 19.443253 20.700000 16.260205
39 52.419385 19.011561 35.872660 33.407825
40 35.492446 11.701672 15.590363 23.790774
49 41.767732 17.899402 20.030886 23.868330
54 41.171680 17.321206 33.469403 23.850474
75 70.221452 39.822724 68.118403 30.398728
83 81.104100 24.794168 77.887020 56.309932
85 45.443750 9.906072 45.000000 35.537678
94 94.174822 15.380770 67.705721 78.794052
95 57.522356 33.647075 50.909858 23.875281
96 96.657315 19.461581 90.211498 77.195734
112 42.021386 -6.554948 67.900000 48.576334
115 129.834041 8.404475 48.384057 121.429566
122 80.074914 48.069531 52.403439 32.005383
141 89.504947 48.903653 72.003423 40.601295
142 85.290173 18.278890 100.744220 67.011283
147 55.080766 -3.759930 56.000000 58.840695
151 48.030624 3.969815 58.344519 44.060809
162 118.144655 38.449501 50.838520 79.695154
163 115.923261 37.515436 76.800000 78.407825
166 56.991404 6.874089 57.009005 50.117315
167 72.343594 16.420790 59.869012 55.922805
168 95.382596 24.822631 95.157633 70.559965
173 50.825029 9.064729 56.300000 41.760300
179 68.721910 49.431864 68.056012 19.290046
180 37.903910 4.479099 24.710274 33.424811
189 82.406524 29.276422 77.054565 53.130102
192 74.469082 33.283157 66.942101 41.185925
197 58.828379 37.577873 125.742385 21.250506
206 95.480229 46.550053 59.000000 48.930176
228 38.046551 8.301669 26.236830 29.744881
235 63.929470 19.971097 40.177050 43.958373
248 47.319648 8.573680 35.560252 38.745967
252 42.918041 -5.845994 58.000000 48.764035
257 50.160078 -2.970024 42.000000 53.130102
263 33.788843 3.675110 25.500000 30.113733
278 40.413366 -1.329412 30.982768 41.742778
287 33.041688 -0.324678 19.071075 33.366366
290 36.422485 13.879424 20.242562 22.543061
304 45.075450 12.306951 44.583177 32.768499
pelvic_radius degree_spondylolisthesis class
7 117.270067 -10.675871 Hernia
10 108.648265 -7.825986 Hernia
11 120.055399 0.499751 Hernia
24 115.577116 -3.237562 Hernia
26 125.203296 -10.093108 Hernia
37 137.540613 -0.263490 Hernia
39 116.559771 1.694705 Hernia
40 106.938852 -3.460358 Hernia
49 118.363389 2.062963 Hernia
54 116.377889 -9.569250 Hernia
75 148.525562 145.378143 Spondylolisthesis
83 151.839857 65.214616 Spondylolisthesis
85 163.071041 20.315315 Spondylolisthesis
94 114.890113 53.255220 Spondylolisthesis
95 140.981712 148.753711 Spondylolisthesis
96 120.673041 64.080998 Spondylolisthesis
112 111.585782 27.338671 Spondylolisthesis
115 107.690466 418.543082 Spondylolisthesis
122 110.709912 67.727316 Spondylolisthesis
141 134.634291 118.353370 Spondylolisthesis
142 110.660701 58.884948 Spondylolisthesis
147 109.915367 31.773583 Spondylolisthesis
151 125.350962 35.000078 Spondylolisthesis
162 81.024541 74.043767 Spondylolisthesis
163 104.698603 81.198927 Spondylolisthesis
166 109.978045 36.810111 Spondylolisthesis
167 70.082575 12.072644 Spondylolisthesis
168 89.307547 57.660841 Spondylolisthesis
173 78.999454 23.041524 Spondylolisthesis
179 125.018517 54.691289 Spondylolisthesis
180 157.848799 33.607027 Spondylolisthesis
189 117.042244 62.765348 Spondylolisthesis
192 146.466001 124.984406 Spondylolisthesis
197 135.629418 117.314683 Spondylolisthesis
206 96.683903 77.283072 Spondylolisthesis
228 123.803413 3.885773 Normal
235 113.065939 -11.058179 Normal
248 120.576972 1.630664 Normal
252 121.606859 -3.362045 Normal
257 131.802491 -8.290203 Normal
263 128.325356 -1.776111 Normal
278 119.335655 -6.173675 Normal
287 120.388611 9.354365 Normal
290 126.076861 0.179717 Normal
304 147.894637 -8.941709 Normal
Visualisasi#
# Visualisasi outlier per fitur (jika ingin diaktifkan)
for col in data1.columns:
plt.figure(figsize=(8, 3))
plt.scatter(range(len(data1[col])), data1[col], label=col)
plt.scatter(
[i[0] for i in outlier_indices if i[1] == col],
data1[col].iloc[[i[0] for i in outlier_indices if i[1] == col]],
color='red', label="Outlier", s=50
)
plt.xlabel("Index")
plt.ylabel(col)
plt.title(f"Outlier Detection for {col}")
plt.legend()
plt.show()
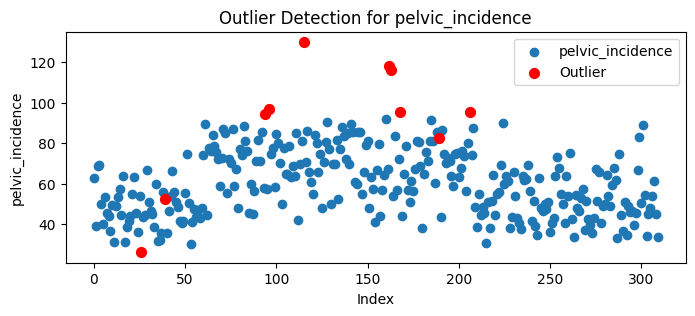
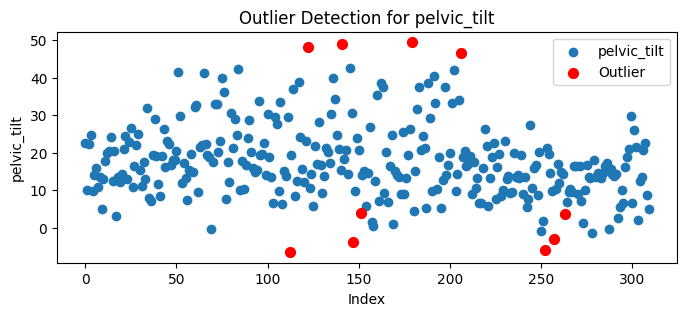
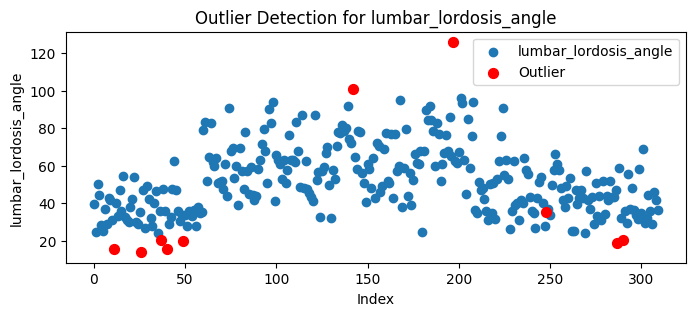
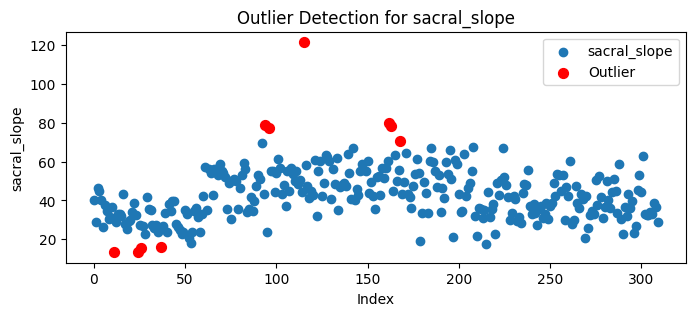
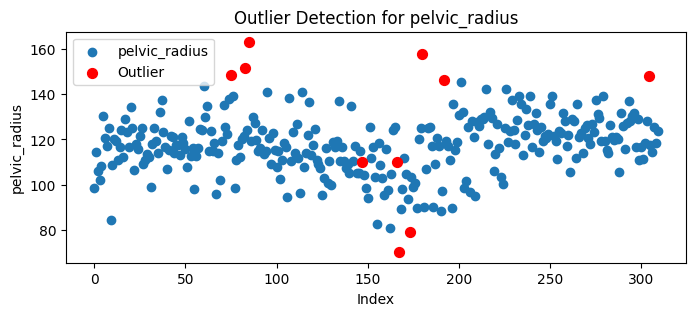

untuk penanganan outlier tidak kita lakukan dikarenakan data yang terindikasi outlier masih berada di rentang yang normal(tidak melebihi interval) dan Distribusi outlier menyebar di semua kelas dan Beberapa nilai negatif pada fitur tertentu
Klasifikasi#
Modelling Klasifikasi Gaussian Naive Bayes#
Modeling merupakan proses pembuatan dan pengujian model statistik atau matematis yang digunakan untuk menggambarkan dan menganalisis pola atau hubungan dalam data. Tujuan utama dari pemodelan dalam data mining adalah untuk mengidentifikasi pola yang berguna atau prediksi yang akurat dari data yang tersedia.Untuk modelling kali ini bertujuan untuk menentukan class pada suatu data inputan .Data akan dibagi menjadi 2 tipe yaitu data test dan data train dan pemodelan kali ini menggunakan metode Gaussian Naive Bayes.
Pada bagian ini akan ditentukan severity dari data yang akan saya inputan apakah termasuk jinak atau ganas.
cara kalkulasi GNB dapat melalui tahap berikut:
Bagi Dataset menjadi data test dan data train
ada dua jenis pembagian rasio dataset yang sering digunakan yaitu:
80% data train dan 20% data test.
70% data train dan 30% data test.
Untuk kali ini kita akan menggunkan raiso 80% data train dan 20% data test,namun kalian bisa mengubahnya tergantung situasi dan kondisi dilapangan nantinya.
Mengghitung Probabilitas pada data train setiap sheet
lakukan perhitungan probabilitas dari masing-masing kelas sesuai dengan jumlah data train pada kelas tersebut kemudian dibagi dengan banyaknya total data train.
ini digunakan untuk menghitung Prior
Menghitung Mean dan Standart dev
hitung mean dan standart deviasi setiap fitur pada setiap kelas di data train kita
menghitung Distribusi gaussian
lakukan perhitungan dengan rumus distribusi gaussian dengan rumus sebagai berikut:
hitung posterior
Setelah didapat semua hasil dari distribusi gaussian, langkah selanjutnya adalah menentukan posteriori-nya. Berikut ini untuk rumusnya:
P(A|Hi) : hasil perkalian setiap fitur pada setiap kelas
P(a) : probabilitas setiap kelas
posteriori = P(A|Hi) * P(a)
tentukan maximum posterior
setelah kita menghitung semua posterior maka dapat kita cari mana yang terbesar
data posterior paling besar tersebut yang merupakan akan menjadi class dari data yang kita inputkan
MODEL
import pandas as pd
from sklearn.naive_bayes import GaussianNB
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import LabelEncoder
from sklearn.metrics import accuracy_score, precision_score, recall_score, f1_score
# Asumsikan data kamu sudah dalam bentuk dataframe "data"
# data = pd.concat([X, y], axis=1) ← sebelumnya sudah dilakukan
# Pisahkan fitur dan label
X = data[['pelvic_incidence', 'pelvic_tilt', 'lumbar_lordosis_angle',
'sacral_slope', 'pelvic_radius', 'degree_spondylolisthesis']]
y = data['class'] # Label kolom
# Encode label jika masih teks
le = LabelEncoder()
y_encoded = le.fit_transform(y)
# Split data menjadi training dan testing
X_train, X_test, y_train, y_test = train_test_split(X, y_encoded, test_size=0.2, random_state=42)
# Buat model Gaussian Naive Bayes
gnb_model = GaussianNB()
# Latih model
gnb_model.fit(X_train, y_train)
# Prediksi
y_pred = gnb_model.predict(X_test)
# Evaluasi
acc = accuracy_score(y_test, y_pred)
prec = precision_score(y_test, y_pred, average='macro') # karena multiclass
rec = recall_score(y_test, y_pred, average='macro')
f1 = f1_score(y_test, y_pred, average='macro')
# Tampilkan hasil
print("========== HASIL EVALUASI NAIVE BAYES ==========")
print(f"Akurasi : {acc:.2f}")
print(f"Presisi : {prec:.2f}")
print(f"Recall : {rec:.2f}")
print(f"F1-Score : {f1:.2f}")
y_pred = gnb_model.predict(X_test)
print("=========================== X TRAINING =================================")
print(X_train)
print("=========================== X TESTING =================================")
print(X_test)
print("=========================== Y TESTING =================================")
print(y_test)
========== HASIL EVALUASI NAIVE BAYES ==========
Akurasi : 0.87
Presisi : 0.83
Recall : 0.82
F1-Score : 0.83
=========================== X TRAINING =================================
pelvic_incidence pelvic_tilt lumbar_lordosis_angle sacral_slope \
126 70.676898 21.704402 59.181161 48.972496
109 68.613001 15.082235 63.014696 53.530766
247 49.828135 16.736435 28.000000 33.091700
234 37.731992 9.386298 42.000000 28.345694
202 76.314028 41.933683 93.284863 34.380345
.. ... ... ... ...
188 85.680950 38.650035 82.680977 47.030914
71 86.900794 32.928168 47.794347 53.972627
106 65.013773 9.838262 57.735837 55.175511
270 51.311771 8.875541 57.000000 42.436230
102 70.399308 13.469986 61.200000 56.929322
pelvic_radius degree_spondylolisthesis
126 103.008354 27.810148
109 123.431174 39.497987
247 121.435558 1.913307
234 135.740926 13.683047
202 132.267286 101.218783
.. ... ...
188 120.840707 61.959034
71 135.075364 101.719092
106 94.738525 49.696955
270 126.472258 -2.144044
102 102.337524 25.538429
[248 rows x 6 columns]
=========================== X TESTING =================================
pelvic_incidence pelvic_tilt lumbar_lordosis_angle sacral_slope \
289 44.430701 14.174264 32.243495 30.256437
9 36.686353 5.010884 41.948751 31.675469
57 46.855781 15.351514 38.000000 31.504267
60 74.377678 32.053104 78.772013 42.324573
25 54.124920 26.650489 35.329747 27.474432
.. ... ... ... ...
198 74.854480 13.909084 62.693259 60.945396
195 71.241764 5.268270 85.999584 65.973493
210 38.505273 16.964297 35.112814 21.540976
224 89.834676 22.639217 90.563461 67.195460
158 57.035097 0.345728 49.198003 56.689369
pelvic_radius degree_spondylolisthesis
289 131.717613 -3.604255
9 84.241415 0.664437
57 116.250917 1.662706
60 143.560690 56.125906
25 121.447011 1.571205
.. ... ...
198 115.208701 33.172255
195 110.703107 38.259864
210 127.632875 7.986683
224 100.501192 3.040973
158 103.048698 52.165145
[62 rows x 6 columns]
=========================== Y TESTING =================================
[1 0 0 2 0 2 2 2 1 0 2 2 1 1 1 1 0 2 2 2 2 2 2 2 2 2 2 0 1 1 2 0 2 1 2 2 0
0 2 2 0 0 2 1 1 2 1 0 1 2 2 1 2 2 2 1 1 2 2 1 1 2]
berikut cara menjadikan data train kita ke dalam bentuk csv.
import pandas as pd
# Concatenate X_train and y_train as new column
train_data = pd.concat([X_train, pd.Series(y_train, name='Severity')], axis=1)
# Save training data to CSV with tab-separated values
train_data.to_csv('Train_mammographic_mass.csv', sep="\t", index=False)
uji coba inputan dengan data test menggunakan model yang kita buat.
# Menampilkan data asli baris ke-231 (baris ke-230 karena index dimulai dari 0)
print("Data yang akan digunakan adalah:")
print(data.iloc[230]) # data adalah DataFrame gabungan X dan y
# Contoh input baru dengan 6 fitur (sesuai struktur data)
# Format: [pelvic_incidence, pelvic_tilt, lumbar_lordosis_angle, sacral_slope, pelvic_radius, degree_spondylolisthesis]
input_data = [[85.0, 20.0, 75.0, 65.0, 120.0, 60.0]]
# Prediksi kelas menggunakan model Gaussian Naive Bayes
predicted_class_index = gnb_model.predict(input_data)[0]
predicted_class_label = le.inverse_transform([predicted_class_index])[0] # kembalikan ke label teks
print("Hasil prediksi:")
print(f"Data yang diinputkan diprediksi sebagai class = {predicted_class_label}")
Data yang akan digunakan adalah:
pelvic_incidence 65.611802
pelvic_tilt 23.137919
lumbar_lordosis_angle 62.582179
sacral_slope 42.473883
pelvic_radius 124.128001
degree_spondylolisthesis -4.083298
class Normal
Name: 230, dtype: object
Hasil prediksi:
Data yang diinputkan diprediksi sebagai class = Spondylolisthesis
/home/codespace/.local/lib/python3.12/site-packages/sklearn/utils/validation.py:2739: UserWarning: X does not have valid feature names, but GaussianNB was fitted with feature names
warnings.warn(
from sklearn.metrics import accuracy_score, precision_score, recall_score, f1_score
# Evaluasi model
accuracy = accuracy_score(y_test, y_pred)
precision = precision_score(y_test, y_pred, average='macro') # rata-rata presisi semua kelas
recall = recall_score(y_test, y_pred, average='macro') # rata-rata recall semua kelas
fscore = f1_score(y_test, y_pred, average='macro') # rata-rata F1 semua kelas
# Menampilkan hasil evaluasi
print("========== EVALUASI MODEL NAIVE BAYES ==========")
print(f'Akurasi : {accuracy:.2f}')
print(f'Presisi : {precision:.2f}')
print(f'Recall : {recall:.2f}')
print(f'F1-Score: {fscore:.2f}')
========== EVALUASI MODEL NAIVE BAYES ==========
Akurasi : 0.87
Presisi : 0.83
Recall : 0.82
F1-Score: 0.83
Modelling Klasifikasi KNN#
K-Nearest Neighbors (KNN) merupakan sebuah cara untuk mengklasifikasikan dengan cara melihat sesuatu yang berada di dekatnya. KNN juga disebut dengan Algoritna oembelajar malas. Karena tidak memerlukan pembelajaran terlebih dahulu, dan langsung meyimpan data set dan pada saat klaifikasi melakukan set data. KNN bekerja dengan menggunakan kedekatan dan pemungutan suara mayoritas untuk membuat prediksi atau forecasting. Pada KNN terdapat istilah “k”. “k” merupakan angka yang memberi tahu algoritma beberapa banya titik terdekat (tetangga) yang digunakan untuk membuat keputusan. Contohnya: Misalkan menntukan nama buah dan membandingkannya dengan buah yang telah dikenal. kemudian menggunakan nilai “k” sebanyak 3 kemudian 2 dari 3 merupakan buah apel dan 1 dari 3 merupakan buah pisang. jadi algoritma ini mengatakan bahwa buah tersebut merupakan apel karena sebagian besar tetangganya apel.
Menentukan Nilai “k”#
Dalam KNN, pemilihan nilai “k” sangat penting untuk menentukan hipotesis hasil dari prediksi. Jika kumpulan data memiliki outlier atau noise yang signifikan, nilai “k” yang lebih tinggi dapat membantu memperhalus prediksi dan mengurangi data yang noise. Namun, pemilihan nilai yang tinggi dapat menyebabkan underfitting.
Pertama, kita harus menentukan nilai K terlebih dahulu. Penentuan nilai K ini tidak ada rumus pastinya. Namun satu tips yang dapat dipertimbangkan, yakni jika kelas berjumlah genap maka sebaiknya nilai K-nya ganjil, sebaliknya jika kelas berjumlah ganjil maka sebaiknya nilai K-nya genap. Dalam prakteknya di Python, Anda dapat menghitung menggunakan kode program untuk mencari nilai K terbaik dari berbagai opsi nilai (misalnya dari K=2 sampai K=10).
Hitung jarak antara data baru dan masing-masing data lainnya#
menghitung jarak menggunakan metode Euclidean distance
Jika ada lebih dari satu, kita dapat menjumlahkannya seperti di bawah ini.
Ambil tiga data dengan jarak terdekat. Dari perhitungan Euclidean distance di atas, jika kita rangkum dari jarak terdekat
Impelementasi KNN menggunakan Scikit Learn#
from sklearn.neighbors import KNeighborsClassifier
from sklearn.metrics import accuracy_score, precision_score, recall_score, f1_score, classification_report
# Gunakan data yang sudah disiapkan sebelumnya:
# X, y_encoded, le → label encoder
# X_train, X_test, y_train, y_test → hasil train_test_split
# Buat model KNN
knn_model = KNeighborsClassifier(n_neighbors=5)
# Latih model
knn_model.fit(X_train, y_train)
# Prediksi
y_pred_knn = knn_model.predict(X_test)
# Evaluasi
accuracy_knn = accuracy_score(y_test, y_pred_knn)
precision_knn = precision_score(y_test, y_pred_knn, average='macro')
recall_knn = recall_score(y_test, y_pred_knn, average='macro')
f1_knn = f1_score(y_test, y_pred_knn, average='macro')
# Tampilkan hasil
print("========== EVALUASI MODEL KNN ==========")
print(f'Akurasi : {accuracy_knn:.2f}')
print(f'Presisi : {precision_knn:.2f}')
print(f'Recall : {recall_knn:.2f}')
print(f'F1-Score: {f1_knn:.2f}')
# Optional: Report klasifikasi
print("\n=== Classification Report KNN ===")
print(classification_report(y_test, y_pred_knn, target_names=le.classes_))
========== EVALUASI MODEL KNN ==========
Akurasi : 0.84
Presisi : 0.78
Recall : 0.77
F1-Score: 0.77
=== Classification Report KNN ===
precision recall f1-score support
Hernia 0.64 0.58 0.61 12
Normal 0.72 0.72 0.72 18
Spondylolisthesis 0.97 1.00 0.98 32
accuracy 0.84 62
macro avg 0.78 0.77 0.77 62
weighted avg 0.83 0.84 0.84 62
Modelling Klasifikasi Decision Tree#
Decision Tree (Pohon Keputusan) adalah algoritma machine learning yang membangun model prediksi dalam bentuk struktur pohon. Algoritma ini memecah dataset menjadi himpunan data yang lebih kecil dan lebih murni secara bertahap, di mana setiap pemecahan didasarkan pada fitur data yang memberikan informasi paling banyak.
Konsep dan Rumus Inti (Decision Tree - C4.5 / ID3)#
1. Entropy (Ukuran Ketidakpastian)#
Entropy digunakan untuk mengukur seberapa acak atau tidak murni suatu set data. Nilai 0 artinya seluruh data dalam satu kelas (murni), sedangkan nilai lebih tinggi artinya campuran berbagai kelas.
\(p_i\) = proporsi jumlah data pada kelas ke-i dari total data.
2. Information Gain (Pengurangan Ketidakpastian)#
Information Gain digunakan untuk menentukan seberapa efektif suatu fitur dalam memisahkan data. Fitur dengan gain tertinggi akan dipilih sebagai node cabang.
Penerapan pada Dataset Vertebral Column#
Dataset ini terdiri dari 3 kelas utama:
Normal
Hernia
Spondylolisthesis
Dan 6 fitur numerik:
pelvic_incidencepelvic_tiltlumbar_lordosis_anglesacral_slopepelvic_radiusdegree_spondylolisthesis
Karena fitur-fitur pada dataset ini bersifat numerik, maka:
Decision Tree akan melakukan pemisahan berbasis nilai threshold, misalnya:
pelvic_incidence <= 65.5 → kiri,> 65.5 → kanan.
Contoh Proses Pembangunan Pohon#
Langkah 1: Menghitung Entropy Awal (Entropy(S))#
Misalkan:
Jumlah data = 310
Distribusi kelas:
Normal = 100
Hernia = 60
Spondylolisthesis = 150
Langkah 2: Menghitung Gain untuk Setiap Fitur#
Misal, untuk fitur pelvic_incidence, data dipecah pada threshold tertentu:
\(S_1\): data dengan
pelvic_incidence <= 65.5\(S_2\): data dengan
pelvic_incidence > 65.5
Hitung entropy masing-masing subset lalu gunakan formula:
Lakukan hal yang sama untuk fitur lain. Fitur dengan Gain tertinggi akan dijadikan node cabang.
Contoh Struktur Pohon Hasil Training (Misal)#
degree_spondylolisthesis <= 25.3?
|
|--- yes ---> class: Normal
|
|--- no ----> pelvic_incidence <= 80.0?
|
|--- yes ---> class: Hernia
|
|--- no ----> class: Spondylolisthesis
Implementasi Decision Tree dengan Scikit-learn#
Setelah kita memahami cara kerja Decision Tree secara manual dengan menghitung Entropy dan Information Gain, kini saatnya kita melihat bagaimana proses ini dilakukan secara otomatis menggunakan library machine learning populer, yaitu Scikit-learn (sklearn). Scikit-learn menyediakan implementasi Decision Tree yang efisien dan siap pakai.
Kita akan menggunakan data yang sudah didiskretisasi sebelumnya, lalu melatih model DecisionTreeClassifier dari Scikit-learn untuk membangun pohon keputusan. Hasil pohon yang dibangun oleh Scikit-learn ini seharusnya konsisten dengan logika perhitungan manual yang sudah kita lakukan.
Terakhir, kita akan memvisualisasikan pohon yang dihasilkan oleh Scikit-learn untuk melihat strukturnya secara grafis.
import pandas as pd
from ucimlrepo import fetch_ucirepo
from sklearn.preprocessing import LabelEncoder
from sklearn.model_selection import train_test_split
from sklearn.tree import DecisionTreeClassifier, plot_tree
from sklearn.metrics import accuracy_score, precision_score, recall_score, f1_score, classification_report
import matplotlib.pyplot as plt
# Fetch dataset dari UCI
vertebral_column = fetch_ucirepo(id=212)
# Ekstrak fitur dan target
X = vertebral_column.data.features
y = vertebral_column.data.targets
# Encode label ('Normal', 'Hernia', 'Spondylolisthesis') menjadi angka
le = LabelEncoder()
y_encoded = le.fit_transform(y)
# Bagi data menjadi train dan test
X_train, X_test, y_train, y_test = train_test_split(X, y_encoded, test_size=0.2, random_state=0)
/home/codespace/.local/lib/python3.12/site-packages/sklearn/preprocessing/_label.py:110: DataConversionWarning: A column-vector y was passed when a 1d array was expected. Please change the shape of y to (n_samples, ), for example using ravel().
y = column_or_1d(y, warn=True)
Training Decision Tree#
# Buat dan latih model Decision Tree
dt_model = DecisionTreeClassifier(criterion='entropy', max_depth=4, random_state=0)
dt_model.fit(X_train, y_train)
# Prediksi
y_pred_dt = dt_model.predict(X_test)
Evaluasi Model Decision TRee#
# Evaluasi kinerja
accuracy_dt = accuracy_score(y_test, y_pred_dt)
precision_dt = precision_score(y_test, y_pred_dt, average='macro')
recall_dt = recall_score(y_test, y_pred_dt, average='macro')
f1_dt = f1_score(y_test, y_pred_dt, average='macro')
# Tampilkan hasil evaluasi
print("========== EVALUASI MODEL DECISION TREE ==========")
print(f'Akurasi : {accuracy_dt:.2f}')
print(f'Presisi : {precision_dt:.2f}')
print(f'Recall : {recall_dt:.2f}')
print(f'F1-Score: {f1_dt:.2f}')
# Tampilkan klasifikasi per kelas
print("\n=== Classification Report ===")
print(classification_report(y_test, y_pred_dt, target_names=le.classes_))
========== EVALUASI MODEL DECISION TREE ==========
Akurasi : 0.76
Presisi : 0.72
Recall : 0.68
F1-Score: 0.67
=== Classification Report ===
precision recall f1-score support
Hernia 0.67 0.31 0.42 13
Normal 0.60 0.79 0.68 19
Spondylolisthesis 0.90 0.93 0.92 30
accuracy 0.76 62
macro avg 0.72 0.68 0.67 62
weighted avg 0.76 0.76 0.74 62
Visualisasi Pohon Keputusan#
# Visualisasi pohon keputusan
plt.figure(figsize=(20,10))
plot_tree(dt_model,
feature_names=X.columns,
class_names=le.classes_,
filled=True,
rounded=True)
plt.title("Visualisasi Pohon Keputusan (Decision Tree)")
plt.show()
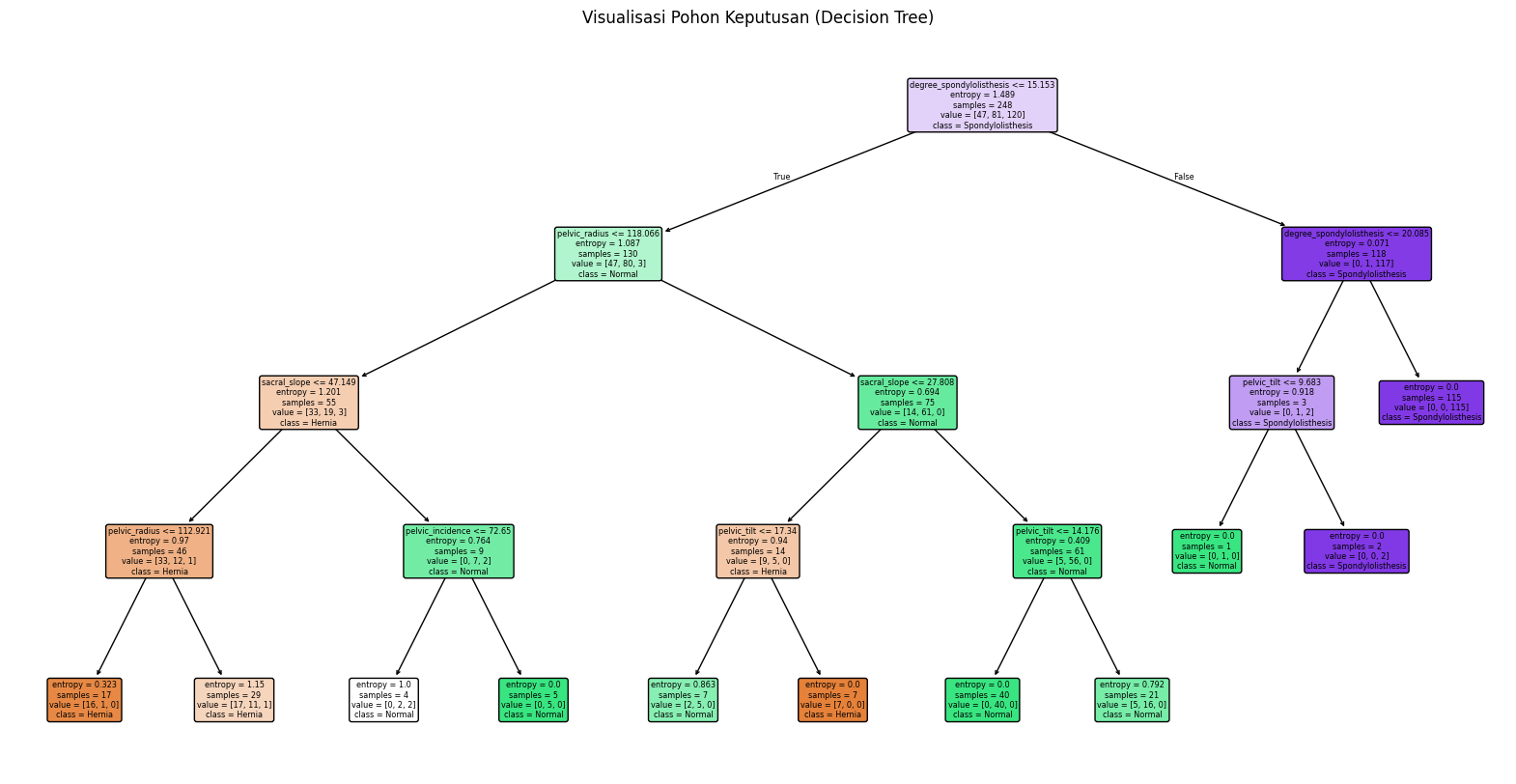
Feature paling berpengaruh: degree_spondylolisthesis, sacral_slope, pelvic_tilt, pelvic_radius.
Model kuat memprediksi Spondylolisthesis, cukup baik untuk Normal, dan perlu perhatian lebih untuk Hernia.
Evaluasi#
Dari ketiga model yang dibuat dapat dilihat akurasi dari masing-masing model, yaitu:
Naive Bayes: memiliki akurasi 0.87
KNN: memiliki akurasi 0.84
Decision Tree: memiliki akurasi 0.76
Dari ketiga model diatas dapat disimpulkan bahwa single model Naive Bayes memiliki akurasi tertinggi yaitu 87%.
